Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de fitopatología
versión On-line ISSN 2007-8080versión impresa ISSN 0185-3309
Rev. mex. fitopatol vol.41 no.3 Texcoco sep. 2023 Epub 13-Oct-2023
https://doi.org/10.18781/r.mex.fit.2210-5
Artículos de revisión
Consideraciones sobre el ARN de interferencia para el control de enfermedades fúngicas en cultivos agrícolas en México y América Latina
1 Unidad de Biotecnología, Centro de Investigación Científica de Yucatán, A.C., Calle 43 No. 130 x 32 y 34, colonia Chuburná de Hidalgo, Mérida C.P. 97205, Yucatán, México;
2 Unidad de Bioquímica y Biología Molecular de Plantas, Centro de Investigación Científica de Yucatán, A.C., Calle 43 No. 130 x 32 y 34, colonia Chuburná de Hidalgo, Mérida C.P. 97205, Yucatán, México;
El control de fitopatógenos es fundamental para la seguridad alimentaria. En la última década se ha propuesto el uso de ARN de interferencia (iRNA) como herramienta tecnológica para el control de enfermedades y plagas agrícolas. Aunque se han descrito distintos enfoques como el uso de “Host-Induce Gene Silencig” (HIGS) y de “Virus-Induce Gene Silencing” (VIGS), más recientemente ha surgido un enfoque no transgénico y amigable con el ambiente, denominado “Spray-Induce Gene Silencing” (SIGS), que hace uso de cadenas dobles de ARN (dsRNA) “desnudos”. Esta revisión analiza trabajos recientes que hacen uso del dsRNA, en especial del SIGS, para el control de hongos fitopatógenos; se enfatizan aspectos de su eficacia, inocuidad en la salud humana y su estabilidad en el medio ambiente. Se enfocan también importantes problemas fitosanitarios en México y América Latina que podrían ser abordadas con el uso del SIGS. La conclusión de esta revisión es que SIGS es una tecnología con verdadero potencial para el control de hongos fitopatógenos en plantas de campo y en frutos postcosecha. Al final se plantean las rutas críticas e investigaciones que deben abordarse para que eventualmente el uso de SIGS pueda concretarse.
Palabras clave: Silenciamiento génico; dsRNA; SIGS; patógenos vegetales
The control of phytopathogens is key for food security. In the last decade, the use of interference RNA (iRNA) has been proposed as a technological tool for controlling diseases and pests in agriculture. Although different approaches have been described, such as the use of “Host-Induce Gene Silencing” (HIGS) and “Virus-Induce Gene Silencing” (VIGS), more recently a non-transgenic and environmentally friendly approach has emerged, called “Spray -Induce Gene Silencing” (SIGS), which uses double-stranded “naked” RNA (dsRNA). This review discusses recent reports on the use of dsRNA, especially SIGS, to control phytopathogenic fungi; emphasizing factors such as efficacy, safety in terms of human health and its stability in the environment. It also focuses on important phytosanitary problems in Mexico and Latin America that can be addressed with SIGS. This review concludes that SIGS technology has real potential to be used to control phytopathogenic fungi on plants in the field and on postharvest fruits. At the end, the critical tasks and the lines of research that must be carried out to promote the SIGS to make it a reality are considered.
Key words: Gene silencing; dsRNA; SIGS; plant pathogens
El constante crecimiento de la población demanda garantizar la producción de alimentos, convirtiéndose en un reto mundial. Se estima que para el 2050 la demanda de alimentos incremente más del 50% (van Dijk et al., 2021), con la responsabilidad además de incrementar la productividad sin provocar mayor impacto ambiental. El desafío es aún mayor, porque las plagas y enfermedades ponen en constante riesgo la seguridad alimentaria. En particular, los hongos fitopatógenos provocan aproximadamente 30% de pérdidas en los cultivos agrícolas en el mundo (Savary et al., 2019). Actualmente el principal método de control es el uso intensivo de fungicidas químicos, ocasionando una fuerte presión de selección sobre los patógenos, favoreciendo mayor diversidad genética y la aparición de aislados más resistentes en sus poblaciones, así como problemas en el medio ambiente y salud humana (Brito et al., 2020). Por estas razones, es cada vez más apremiante proteger los cultivos sin depender de fungicidas químicos. Una opción que ofrece grandes oportunidades es el uso del ARN de interferencia (iRNA), sobre lo cual se tratará la presente revisión.
El iRNA es un mecanismo conservado, presente en la mayoría de las células eucariotas, que se relaciona con la represión de la transcripción (regulación transcripcional de la expresión génica) y/o degradación del ARN mensajero (mRNA), o inhibición de la traducción (regulación post-transcripcional); también participa en la defensa de los organismos contra elementos extraños (Garcia-Ruiz et al., 2016; Rosa et al., 2018). Este mecanismo, conocido como silenciamiento génico, es desencadenado por estructuras de ARN de doble cadena (siglas en inglés dsRNA, por “double strand RNA”), que pueden tener una estructura de dúplex lineales o una estructura tipo “hairpin” (horquilla, también llamada estructura tallo-asa) (Figura 1). Los sRNAs (por “small RNA”, o ARN pequeños) que desencadenan el silenciamiento pueden dividirse en dos grandes grupos según su origen, los micro-ARNs (miRNA) y los pequeños ARN de interferencia (siRNA, de small interfering RNA).

Figura 1 Mecanismos de producción de los ARN pequeños (sRNA) en eucariotas. Los sRNA comprenden los pequeños ARN de interferencia (siRNA) y los micro ARNs (miRNA). En la izquierda se presentan las diferentes rutas de biosíntesis de los siRNA, los cuales pueden tener la forma estructural de doble cadena lineal o de hairpin (horquilla o tallo-asa). Estas dos estructuras (dúplex lineal y hairpin) son las estructuras que imitan los sRNA de doble cadena (dsRNA) artificiales, es decir, diseñados con fines biotecnológicos. A la derecha se observa la biosíntesis de los miRNA que provienen de los genes MIR, a partir de los cuales se transcriben los pri-RNA. El silenciamiento inicia cuando los pri-RNA o los precursores de los siRNA son reconocidos y cortados por DCL (proteína Dicer), generando los siRNA y los pre-miRNA respectivamente. Estos últimos son nuevamente cortados por DCL a miRNA. Los siRNA y miRNA posteriormente se unen a la proteína AGO (argonauta), formando el complejo RISC (por “RNA-induced silencing complex” o complejo de silenciamiento inducido por ARN). En RISC, una hebra del siRNA o del miRNA es usada para guiar el silenciamiento transcripcional o post transcripcional. En hongos el sistema de silenciamiento regula, tanto a nivel transcripcional como post transcripcional, procesos de desarrollo, defensa y patogenicidad.
Los miRNA provienen de la transcripción de los genes MIR; el transcrito inicial es llamado pri-miRNA y forma una estructura de horquilla que es reconocida por una ribonucleasa III tipo Dicer (DCL, por Dicer-like), que los corta a estructuras más pequeñas llamadas pre-miRNA. DCL hace un nuevo corte sobre los pre-RNA y se producen los miRNA.
Los siRNA por su parte, son producidos a través de la transcripción de secuencias repetidas o de elementos de transposición, o por la replicación en el genoma de secuencias de origen viral, o por la amplificación secundaria de dsRNA después de que los sRNA fueron cortados por Dicer, o por hibridaciones de regiones complementarias entre dos transcritos independientes (que codifican diferentes proteínas). Los dsRNA originales, producidos por cualquiera de estas vías, son procesados por DCL en fragmentos más pequeños.
Ambos sRNA (miRNA y siRNA), son fragmentos de aproximadamente 19-24 nucleótidos (nt), y son reclutados por la proteína Argonauta (AGO) y procesados en el complejo de silenciamiento inducido por ARN (RISC, por “RNA-induced silencing complex”). El complejo RISC usa una de las dos cadenas del sRNA para guiar la degradación específica del mRNA diana o blanco, con el cual el sRNA es complementario; de este modo el sistema regula la expresión de genes de una manera específica a la secuencia. La Figura 1 esquematiza la producción y procesamiento de ambos tipos de sRNAs: miRNA y siRNA.
Las plantas tienen la habilidad natural de intercambiar sRNAs con sus patógenos, en un proceso denominado “Crosskingdom iRNA” o intercambio cruzado entre reinos, con lo cual las plantas pueden combatir a los patógenos (Hudzik et al., 2020) (Figura 2). En la biotecnología, existen tres enfoques para aprovechar el iRNA para proteger los cultivos: 1) la expresión de transgenes en la planta, llamado HIGS por “host-induced gene silencing” (silenciamiento génico por la producción de dsRNA por el hospedante). HIGS es una estrategia para silenciar los genes de los patógenos a través de la producción in vivo de sRNA complementarios a las secuencias del patógeno, mediante el uso de la ingeniería genética y a través de la transformación con Agrobacterium tumefaciens, resultando en una planta que es un organismos genéticamente modificado u OGM (Halder et al., 2022; Ghag et al., 2019; Koch y Wassenegger, 2021). La mayoría de los genes que han sido dianas en los hongos han sido genes estructurales, reguladores del desarrollo y factores de patogenicidad (Yin y Hulbert, 2015). 2) uso de vectores virales recombinantes, que al replicarse producen los sRNA en las plantas, dirigidos contra un fragmento del transcrito diana y de esta manera desencadena su silenciamiento. Este procedimiento se conoce como VIGS, por “virus-induced gene silencing” (silenciamiento génico por dsRNA producidos con base en un vector viral) (Villanueva-Alonzo et al., 2022). Un ejemplo es el uso del Virus del mosaico estriado de la cebada (Barley stripe mosaic virus-BSMV) para el silenciamiento de genes importantes del hongo Puccinia striiformis f. sp. tritici y evitar la infección del trigo (Triticum aestivum) (Qi et al., 2018; Yin et al., 201). 3) la aplicación exógena de dsRNA, homólogos a genes exclusivos del patógeno. A este método se le conoce como SIGS, por “spray induced gene silencing”, o silenciamiento génico inducido por aspersión de dsRNA (Wang y Jin, 2017). Tanto HIGS como VIGS implican el manejo de organismos genéticamente modificados (OGMs); el primero porque requiere la transformación genética de las plantas de interés y el segundo por la modificación genética de los virus, con los constructos de expresión, para replicarse en las células vegetales y transcribir los sRNA (Figura 3). El Cuadro 1 muestra un resumen de la comparación de las tres técnicas, así como las ventajas y desventajas de cada una. En varios países las regulaciones del uso de OGMs en el campo aun es restringido, lo que abre una ventana de oportunidad para el SIGS, ya que los dsRNA aplicados exógenamente son transitorios y no requiere crear o aplicar OGMs en el campo. El objetivo de este manuscrito es presentar el potencial del SIGS para convertirse en la vía de uso de los iRNA en el campo para el manejo de hongos fitopatógenos, en particular en México y en Latinoamérica, y los aspectos que faltan superar para que esta tecnología pueda usarse exitosamente en el campo.

Figura 2 Representación global del movimiento de sARNs y sus funciones entre el hongo y el hospedante. Los sRNA vegetales son enviados a través de vesículas extracelulares hacia la célula fúngica para silenciar genes involucrados en su crecimiento o virulencia. Los sRNA fúngicos promueven la virulencia y el crecimiento del hongo, y también bloquean la respuesta vegetal de defensa. Este proceso de intercambio de sRNAs entre patógenos y hospedantes es llamado “Crosskigdom iRNA” (interferencia entre reinos mediado por ARN). Cuando los sRNA de doble cadena (dsRNA) exógenos (diseñados contra dianas importantes, generalmente involucrados en el crecimiento fúngico y/o su virulencia), son asperjados directamente sobre el hongo o sobre la hoja, son absorbidos por el hongo, dando lugar a siRNA que bloquean los genes diana que se seleccionaron. La figura muestra también que el silenciamiento artificial (diseñado con fines biotecnológicos), ocurre en el patosistema al mismo tiempo que el silenciamiento de genes del hospedante por envío de sRNAs naturales por parte del hongo, y también silenciamiento de genes del hongo por envío de sRNAs naturales por parte del hospedante.
Cuadro 1 Comparación de tecnologías basadas en ARN de interferencia para el control de plagas y enfermedades agrícolas
| Tecnología | Significado del acrónimo | Descripción | Ventajas | Desventajas | Referencias |
|---|---|---|---|---|---|
| HIGS | Host Induced Gene Silencing (silenciamiento génico por la producción de dsRNA por el hospedante). | La planta es transformada con un vector de ADN que integra el constructo de silenciamiento al genoma. | Producción estable y permanente de los dsRNA. Los dsRNA tienen mayor probabilidad de absorción por los patógenos de diferentes reinos. El procesamiento de los dsRNA depende de la maquinaria de silenciamiento de la planta. | Requiere protocolos de transformación y regeneración a planta de la especie que se quiera convertir en HIGS. La planta es OGM. Poca acceptación de consumidores. | Halder et al., 2022; Papadopoulou et al., 2020; Wang et. al., 2016 |
| VIGS | Virus Induced Gene Silencing (silenciamiento génico por dsRNA producidos con base en un vector viral). | La planta es inoculada con el vector de silenciamiento viral que contiene un fragmento del gen diana. El nuevo ADN no se integra al genoma vegetal. | No requiere de un protocolo de transformación de la planta de interés. El vector VIGS se inocula por bombardeo o agroinfiltración. El procesamiento de los dsRNA depende de la maquinaria de silenciamiento de la planta. | Requiere que el vector VIGS sea capaz de moverse, replicarse y transcribirse en la planta de interés. La producción de los dsRNA es temporal. No provee de una resistencia permanente hacia los patógenos. El vector viral es un OGM. | Halder et al., 2022; Villanueva-Alonzo et al., 2022 |
| SIGS | Spray Induced Gene Silencing (silenciamiento génico inducido por dsRNA asperjados). | Los dsRNAson producidos por transcripción in vitro o sintetizados químicamente. | Para la transcripción in vitro si se generan constructos de silenciamiento, pero los OGMs solo se manejan en el laboratorio. Las plantas asperjadas con dsRNA no son OGMs. | Los dsRNA entran de manera ineficiente a la planta y se degradan fácilmente sobre la superficie vegetal. La producción in vitro actualmente es costosa. El éxito del silenciamiento depende de que el patógeno posea una maquinaria de silenciamiento eficiente. | Gurusamy et al., 2020a, 2020b; Hoffle et al., 2020; Koch y Wassenegger, 2021; Qiao et al., 2021; Sarkar y Roy-Barman, 2021 |
En los casos de HIGS y VIGS, para el procesamiento de los dsRNA (es decir, el corte de los dsRNA a siRNA por Dicer, el reconocimiento de los siRNA por Argonauta y la formación del complejo RISC), depende casi totalmente de la maquinaria de la planta (Höfle et al., 2020), mientras que en el SIGS depende de la capacidad del patógeno de absorber y procesar los dsRNA en siRNA (Koch y Wassenegger, 2021; Qiao et al., 2021); por tanto, para el éxito del SIGS se requiere una maquinaria funcional en el microorganismo diana. En consecuencia, es importante analizar los componentes de la maquinaria de silenciamiento de los hongos.
Maquinaria de silenciamiento en los hongos
Como se ha mencionado previamente, el sistema de silenciamiento es conservado en los eucariotas. En los primeros reportes realizados en Neurospora crassa y Schizosaccharomyces pombe, el silenciamiento recibió el nombre de “quelling” (sofoco o apagamiento de la expresión génica). Así, las proteínas fúngicas relacionadas al silenciamiento recibieron los nombres de QDE, por “quelling defective”. Pero estas denominaciones pueden ser confusas porque mientras QDE1 es una ARN polimerasa dependiente de ADN, QDE2 es una proteína tipo argonauta. Similarmente, SMS2 (por “Suppressor of meiotic silencing 2”), es una proteína homóloga a argonauta en Neurospora crassa (Gaffar et al., 2019). Aunque en las narrativas sobre las primeras investigaciones se siguen mencionando a las proteínas QDE, la tendencia es nombrar a las proteínas del sistema de silenciamiento como sus homólogas en plantas, es decir como dicer (DCL), argonauta (AGO), y ARN Polimerasas dependientes de ARN (RDRP, por RNA-dependent RNA polymerase). En los hongos el número de miembros descritos en estas familias son menores a las descritas en las plantas. Por ejemplo, en el reino vegetal se han descrito más de 15 miembros en la familia AGO y al menos 6 RDRP (García-Ruiz et al., 2016); mientras que en los hongos se conocen tres miembros de AGO, dos DCL y cinco helicasas tipos SAD (por “suppressor of ascus”; supresor de la formación de las ascas) (Lax et al., 2020).
El silenciamiento génico en los hongos, como en otros eucariontes, participa en múltiples procesos (Figura 1) como son la regulación de la expresión genética, la formación de heterocromatina, la defensa contra virus, el control de elementos transponibles, la adaptación a condiciones de estrés, la división celular y desarrollo, y también en la patogénesis (Lax et al., 2020; Zanini et al., 2021). Recientemente, Gaffar y colaboradores (2019) al estudiar los procesos de conidiación, ascoporogénesis, virulencia y producción del metabolito deoxinivalenol en Fusarium graminearum observaron diferentes combinaciones de AGO, DCL y RDRP en los diferentes procesos, lo que evidencia la complejidad del mecanismo de silenciamiento a pesar de que en los hongos se conocen pocos miembros en las principales familias de las proteínas involucradas.
Papel del ARN de interferencia en la virulencia fúngica
El silenciamiento juega un papel importante en la virulencia de los hongos (Gaffar et al., 2019; Weiberg et al., 2013; Yin et al., 2020; Zanini et al., 2021), por lo que estas proteínas se han propuesto como dianas para el control de patógenos vegetales (Haile et al., 2021; Werner et al., 2020). Por ejemplo, Botrytis cinerea es capaz de liberar sRNAs (Bc-sRNAs) durante la infección de las células de sus hospedantes (Cai et al., 2018). Estos Bc-sRNAs actúan secuestrando a la proteína AGO del hospedante, por ejemplo de Arabidopsis thaliana o Solanum lycopersicum, la cual es pieza clave en el sistema de defensa en ambos hospedantes (Weiberg et al., 2013). Interesantemente, un solo sRNA, nombrado Bc-siR37, suprime al menos ocho genes involucrados en la defensa de A. thaliana. Entre ellos, factores de transcripción tipo WRKY, cinasas tipo receptores y enzimas modificadoras de la pared celular (Wang et al., 2017). Verticillium dahliae es otro patógeno en el que se ha estudiado el papel de los sRNA en la invasión del hospedante, y se encontró que usa mecanismos parecidos al descrito en B. cinerea (Wang et al., 2016). En otro ejemplo, Fusarium graminearum también libera Fg-sRNAs, los cuales le permiten silenciar genes de defensa en los hospedantes, favoreciendo la colonización (Jian y Liang, 2019; Werner et al., 2021). Cuando F. graminearum invade trigo (T. aestivum), secreta el sRNA Fg-sRNA1, el cual reduce la expresión de una proteína que posee dominio de unión a quitina (“chitin elicitor binding protein”, TaCEBiP) que posee función elicitora de la defensa (Jian y Liang, 2019). Adicionalmente, se ha comprobado que los Fg-sRNAs dependientes de DCL, regulan en general la expresión de los genes de defensa en las gramíneas (Werner et al., 2021). La eliminación de varios componentes del sistema de producción de iRNA, en particular DCL1 y AGO2, tiene como resultado una disminución del tizón en espigas de trigo, resaltando la importancia de estas proteínas en la virulencia de F. graminearum (Gaffar et al., 2019). Por esa razón, ambos genes (DCL1 y AGO2) han sido propuestos para el control de este patógeno en hojas de cebada (H. vulgare) (Werner et al., 2020). En Penicillium italicum en cambio, la proteína DCL2 es la que regula la expresión de los microRNAs, y contribuye de manera crucial en la patogénesis (Yin et al., 2020), lo que evidencia que la importancia de las diferentes DCL puede variar entre especies; cabe aclarar que en el caso de los hongos los miRNAs reciben el nombre de milRNAs (por micro-RNA like o parecidos a los micro-ARNs). Recientemente, Haile y colaboradores (2021) construyeron una quimera para silenciar la expresión de ambos genes DCL en Plasmopara viticola, estrategia que logró suprimir la capacidad de este patógeno para colonizar la vid (Vitis vinifera).
En el hongo Valsa mali la virulencia se regula con un milRNA nombrado VdmilR1, que opera a nivel transcripcional sobre el extremo 3’ UTR de VdHy1 a través de la metilación K9 de la histona H3; VdHy1 es necesaria para la virulencia de V. mali sobre plantas de Gossypium herbaceum (algodón) (Jin et al., 2019). Interesantemente, a diferencia de otras rutas canónicas de biogénesis de milRNAs, la biogénesis de VdmilR1 no depende de DCL ni de AGO, pero si de una proteína llamada VdR3 que contiene un dominio de RNAsa III (Jin et al., 2019), lo que pone en relieve la importancia de caracterizar en particular el patosistema que se quiere controlar. Igualmente, los milRNAs juegan un papel fundamental en la regulación post-transcripcional de genes de virulencia en V. mali durante la infección en Malus domestica (manzana) (Xu et al., 2020), así como en la desestabilización del sistema de defensa del hospedante (Xu et al., 2022). Estos datos sugieren que la maquinaria de silenciamiento en su conjunto provee un mecanismo conservado en los hongos, para la invasión de sus hospedantes (Figura 2). Los sRNA en su mayoría tienen como objetivo inhibir genes relacionados al sistema de defensa vegetal, y funcionan parecido a las proteínas efectoras de los patógenos (Todd et al., 2023).
Posibilidades del arn de interferencia en la agricultura
Se ha propuesto que el uso del iRNA puede representar una poderosa herramienta tecnológica para el control de enfermedades fúngicas de plantas (Wang y Jin, 2017; Zotti et al., 2018), y otros patógenos como virus, insectos y nematodos (Koch y Wassenegger, 2021). En el caso de HIGS, la mayoría de los trabajos se basan en el uso de vectores binarios que sobreexpresan secuencias invertidas, separados por un intrón, que permite la formación de la horquilla o “hairpin” (Figura 3). Sin embargo, este tipo de enfoques está restringido a plantas en las que existen métodos de transformación establecidos, y para la mayoría de las plantas de interés agronómico no se cuenta con estos protocolos (Halder et al., 2022; Wang et al., 2016). La mayor limitante en muchos países es el uso inherente de OGMs, debido la limitada aceptación y falta de regulaciones para su uso (Papadopoulou et al., 2020). En el caso de VIGS, usa vectores virales de ARN o ADN que tienen la capacidad de transcribir genes foráneos, sin tener que transformar a la planta completa (Halder et al., 2022; Villanueva-Alonzo et al., 2022). Este enfoque está limitado a la disponibilidad y eficiencia de vectores virales para la planta de interés, pero también implica el manejo de genomas virales manipulados genéticamente (Figura 3). Por su parte, en la tecnología de SIGS los dsRNA se aplican de forma exógena sobre la superficie del tejido vegetal, sin necesidad de transformación (Koch et al., 2016; Sarkar y Roy-Barman, 2021). Para el uso de SIGS, los dsRNA se producen mediante la síntesis química, de cada cadena independiente (sentido y antisentido), las cuales luego se mezclan de manera equimolar, es decir en las mismas cantidades, para dar lugar a la doble cadena. La síntesis química de cada cadena es análoga a la síntesis comercial de oligonucleótidos, pero empleando ribonucleótidos como bloques de construcción para la síntesis de las cadenas de ARN. Los dsRNA también pueden producirse mediante transcripción in vitro. En este caso la secuencia que se seleccionó para silenciar en el transcrito diana, se clona en un vector de ADN circular, bajo la regulación de un promotor de origen bacteriófago, como T3, T7 o SP6. Se generan dos clonas, una con la secuencia en sentido (dirección 5´-3´), y la otra con la secuencia en antisentido (dirección 3´-5´). Para la transcripción, ambos vectores son digeridos con una enzima de restricción que tiene un solo sitio de corte en estos vectores y éstos se abren y quedan linealizados. La transcripción in vitro se realiza en un tubo, con una mezcla de reacción que contiene los vectores linealizados, una ARN polimerasa que reconozca al promotor que se esté usando (ya sea T3, T7 o SP6), y los ribonucleótidos (Li y Zamore, 2019; Sun y Riggs, 2017). La Figura 4A muestra cómo se sintetizan in vitro los dsRNA.

Figura 3 Representación esquemática de los diferentes enfoques de control usando el mecanismo de RNA de interferencia. HIGS (host induced gene silencing; es silenciamiento génico mediado por sRNA producidos en el hospedante); en HIGS el hospedante es transformado y modificado genéticamente para la generación de sRNA artificiales endógenos; el casete de silenciamiento es introducido mediante Agrobacterium tumefasciens con un vector de tipo T-DNA en el que se clonan los fragmentos sentido (barra amarilla con flecha a la derecha) y antisentido (barra verde, con flecha a la izquierda) flanqueando una secuencia independiente (representada con un rectángulo negro). Las regiones amarillo y verde son las mismas secuencias, pero invertidas una respecto a la otra, por lo que son complementarias. En el casete de expresión, P corresponde al promotor y T al terminador. Al transcribirse el dsRNA, se forma una estructura tipo hairpin. En VIGS (virus induced gene silencing; silenciamiento génico por dsRNA producidos por vectores virales), el virus es genéticamente modificado para la generación de cada una de las hebras del sRNA (sentido y antisentido). SIGS (spray induced gene silencing), es el silenciamiento génico por dsRNA asperjados exógenamente.

Figura 4 SIGS para el control de hongos, virus e insectos. A) Producción in vitro de los dsRNA para la aspersión exógena. El fragmento diana es clonado en un vector circular bajo la regulación del promotor de un bacteriófago (T3, T7 o SP6); se genera un vector para la cadena sentido y otro para la cadena antisentido. Para la transcripción in vitro, el vector es linealizado con una enzima de restricción que corta una sola vez el vector (sitio único), y ese templado se mezcla con los ribonucleótidos y la ARN polimerasa (T3, T7 o SP6 ARN polimerasa, según sea el promotor usado), que lleva a cabo la síntesis in vitro del dsRNA. En el casete de expresión, P corresponde al promotor y T al terminador. Se sintetizan las dos hebras (sentido y antisentido) que forman el dsRNA. B) El uso de SIGS para el control de patógenos y plagas en frutas y verduras en condiciones de post cosecha es una de las aplicaciones que se puede lograr más pronto.
SIGS tiene ventaja regulatoria, ya que los cultivos asperjados con los dsRNA no están considerados como OGMs (Dalakouras et al., 2020; Taning et al., 2020). Sin embargo, también existen limitantes que tendrán que superarse para la aplicación de SIGS en el campo.
Aplicaciones tópicas en plantas no modelo y productos cosechados
El uso de SIGS es una alternativa a los trabajos clásicos de transformación genética. Este enfoque se ha descrito como amigable con el medio ambiente, altamente específico y con un amplio espectro de cultivos en los que se pudiera aplicar. Es importante mencionar que puede ser aplicado tanto a los cultivos en el campo, como en los productos agrícolas ya cosechados (Figura 4B).
Los primeros análisis en cuanto a su factibilidad se realizaron en plantas modelo como Nicotiana benthamiana y A. thaliana (Dalakouras et al., 2018; Wang et al., 2016), pero también se han evaluado en cebada (H. vulgare), arroz (Oryza sativa), canola (Brassica napus), y cucurbitáceas (Haile et al., 2021; Kaldis et al., 2018; Qiao et al., 2021; Sarkar y Roy-Barman, 2021); en frutos cosechados de fresa (Fragaria vasca), vid (Vitis vinifera), jitomate (S. lycopersicum) y manzana (M. domestica) (Qiao et al., 2021; Wang et al., 2016); y vegetales como lechuga (Lactuca sativa) y cebolla (Allium cepa) (Qiao et al., 2021) y flores (Qiao et al., 2021; Wang et al., 2016). Se ha observado que la aplicación tópica de los dsRNA otorga protección no solo en el sitio de aplicación, sino en otras partes de la planta. La capacidad de las plantas para absorber dsRNA a través de las hojas varía, pero existen alternativas como la aplicación en los peciolos (Dalakouras et al., 2018). Estudios con Sclerotinia sclerotiorum sugieren que la absorción de las dsRNA en los hongos ocurre predominantemente en la punta de la hifa, a través de endocitosis mediado por la proteína clatrina (Wytinck et al., 2020). Los resultados de todas estas recientes investigaciones muestran el potencial de los dsRNA para el control de enfermedades fúngicas en cultivos agrícolas.
Ventajas y desventajas de los dsRNA en la protección vegetal
Especificidad de los dsRNA. La gran ventaja de emplear dsRNA es que se puede decidir silenciar un gen o una familia génica, o varios genes diferentes de un organismo, donde se puede disminuir dramáticamente la virulencia o incluso evitar el crecimiento del hongo. También, puede optarse por silenciar el gen de un solo organismo, o de múltiples organismos a la vez (Haile et al., 2021; Koch et al., 2019; Nerva et al., 2020; Werner et al., 2020). La especificidad de los dsRNA depende de la secuencia per se empleada (Wang et al., 2016). Algunos genes fúngicos son monogénicos, es decir, están solo una vez en el genoma, por ejemplo, el gen LYS2 que codifica a la enzima a-aminoadipato reductasa, o el gen HGMR, que codifica a la enzima hidroxi-metil-glutaril CoA reductasa. En estos casos el silenciamiento es sobre un solo gen. Otros genes tienen múltiples miembros en el genoma y se dice que forman una familia génica; por ejemplo, los genes de catalasas, de quitinasas, de cinasas, etc. Para el diseño del silenciamiento se comparan las secuencias de los ARN mensajeros y se puede elegir alguna región que es idéntica entre todos los miembros de la familia y silenciarlos todos, o silenciarlos por grupos, o seleccionar regiones que son diferentes en cada uno y se silencia un miembro específico (Figura 5), ya sea para probar su relevancia dentro de la familia génica, o porque es el que contribuye a la patogenicidad del hongo. Para lograr la especificidad y evitar dianas no deseadas, se aconseja dirigir el diseño de silenciamiento hacia regiones no conservadas, ya sea dentro de la región codificante o en los extremos 5´y 3´UTR del mRNA (Untraslated Region; región no traducible), sobre todo en el caso de genes que codifican proteínas altamente conservadas. Los UTRs forman parte del ARN mensajero y se localizan respectivamente en los extremos de éste; son importantes para su estructura, pero no son traducidos a aminoácidos. Aun en genes de proteínas altamente conservadas, las secuencias de los UTR varían.
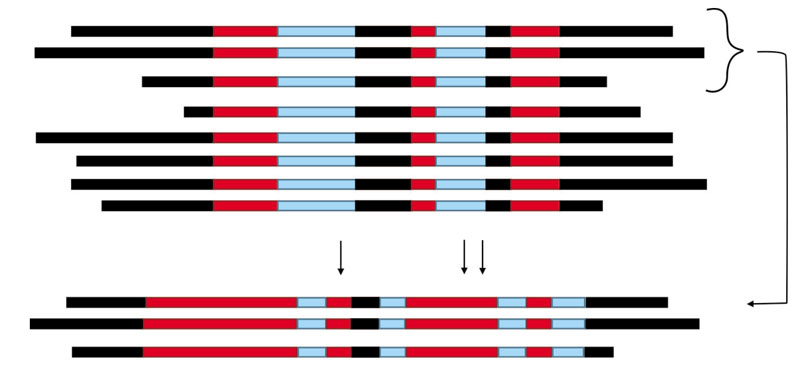
Figura 5 Estrategias para diseñar dsRNA para el silenciamiento específico o múltiple de genes. Para ello, se comparan las secuencias de nucleótidos mediante alguna herramienta bioinformática para alineamientos múltiples. El esquema puede representar diferentes situaciones: A) el mismo gen, pero las secuencias son de diferentes organismos. B) los diferentes genes de una familia génica de un mismo organismo (por ejemplo, la familia de las catalasas, porque hay múltiples genes en el genoma de un organismo). Las barras rojas representan las regiones altamente conservadas, incluso al 100%; las barras azules corresponden a regiones moderadamente conservadas, y las negras a las regiones más divergentes. Los dsRNA diseñados sobre las regiones rojas serían capaces de silenciar el gen en diferentes organismos (situación A) o a todos los miembros de la familia génica (situación B) en un organismo (dsRNA no específicos). Los dsRNA diseñados sobre alguna de las secuencias en negro producirán silenciamiento específico, ya sea del gen de un organismo sin afectar a otros (situación A), o de un solo gen en una familia génica en un organismo (situación B) (dsRNA específico). El alineamiento de abajo puede representar: C) un gen en un grupo de organismos filogenéticamente muy cercanos, o D) los miembros de una subfamilia génica; se observa que algunas regiones que eran azules en el alineamiento global (de arriba), en los organismos más cercanos (situación C) o la subfamilia génica (situación D) son altamente conservadas (en rojo), y permiten diseñar dsRNA que silencian a ese grupo de organismos, sin afectar a otros organismos (situación C), o todos los miembros de la subfamilia, pero sin silenciar otros miembros de esta familia génica (situación D).
La estrategia que se sigue es similar para aplicar el silenciamiento sobre un solo organismo, o múltiples especies de organismos, un género particular, etc.; es decir, depende si se escogen regiones que están altamente conservadas entre todos ellos, o secuencias que son exclusivas de un organismo particular. Por ejemplo, Gutiérrez-Domínguez et al. (2022) reportaron recientemente un tipo de lipasa que en hongos filamentosos sólo está presente en algunos géneros de los órdenes Hypocreales y Glomerellales; estos incluyen los géneros Trichoderma, Fusarium, Nectria, y Colletotrichum. Si se eligen regiones altamente conservadas en el gen de esta lipasa, el silenciamiento ocurriría en todos estos hongos, pero si se eligen secuencias que divergen entre ellas, el silenciamiento puede dirigirse únicamente a las que se quiere controlar, para no afectar por ejemplo a las especies del género Trichoderma, que son hongos benéficos para la agricultura, mientras los otros son fitopatógenos.
La disponibilidad de bases de datos públicas de genomas y transcriptomas, las cuales están en constante crecimiento, permiten diseñar correctamente las estrategias de silenciamiento y evitar que éste ocurra en organismos que no son diana, es decir, que no se quieren afectar.
dsRNA, de bajo riesgo para humanos. De acuerdo a Fletcher et al. (2020), Jensen et al. (2013), y Rodrigues y Petrick (2020), el riesgo por consumir dsRNA en humanos es bajo o nulo. Para que los dsRNA aplicados en frutos o plantas tengan un posible impacto sobre la salud humana, los dsRNA tienen que internarse dentro de las células y coincidir con la secuencia de alguno(s) de los genes humanos (Fletcher et al., 2020). Sin embargo, en el humano y mamíferos en general, hay una multitud de barreras biológicas en el tracto gastrointestinal, torrente sanguíneo y a nivel celular, en los que se degradan por la gran cantidad de nucleasas existentes (Fletcher et al., 2020; Rodrigues y Petrick, 2020). Adicionalmente, el hombre ha consumido de manera natural dsRNA de origen vegetal, a través de la dieta convencional en el maíz (Zea mays), soya (Glycine max), arroz (O. sativa), lechuga y jitomate (Jensen et al., 2013). Para la supresión del gen humano se requeriría que los sRNA fueran complementarios y estuvieran acumulados en concentraciones biológicamente importantes en el sitio blanco, lo cual no ocurre (Jensen et al., 2013).
No persistencia de los dsRNA en la planta. En un trabajo pionero de Koch y colaboradores (2016), aplicaron en hojas de H. vulgare (cebada) dsRNA diseñados contra transcritos de la proteína GFP (green fluorescent protein; proteína verde fluorescente) y 48 h después infectaron las hojas con una cepa de F. graminearum transformada, que produce la proteína GFP. Interesantemente, el silenciamiento de GFP se mantuvo durante 6 días post infección (dpi) en regiones distales de donde se aplicó el dsRNA-GFP, lo que muestra su estabilidad en el tejido y que el dsRNA se mueve dentro de la planta; similarmente, la aplicación en rosas (Rosa chinensis) y frutos de jitomate (S. lycopersicum) de dsRNA que silencian la DCL1/2 de B. cinerea los protegió contra este patógeno por 8 -10 días. Reportes en otras plantas, con otros patógenos fúngicos, coinciden con estos tiempos de estabilidad de los dsRNA (Nerva et al., 2020; Sarkar y Roy Barman, 2021).
En varios trabajos reportan la aplicación primero de los dsRNA y luego inoculan con el patógeno (Höfle et al., 2020; Koch et al., 2016; Koch et al., 2019; Werner et al., 2020; Sarkar y Roy-Barman, 2021); es decir, un efecto preventivo de la infección. Recientemente, Haile y colaboradores (2021) infectaron hojas de Vitis vinifera con Plasmopara viticola, dejaron progresar la enfermedad por 7 días, y posteriormente aplicaron los dsRNA. Estos lograron reducir la tasa de progreso de la enfermedad, implicando que el control con dsRNA pudiese ser también de tipo curativo. Sin embargo, los dsRNA aplicados de manera exógena son extremadamente susceptibles a la degradación y tienen muy poca persistencia en el ambiente (Bachman et al., 2020), por lo que uno de los principales retos para su éxito en la agricultura es alargar su estabilidad en el ambiente.
Factores que afectan el diseño de dsRNA para SIGS. Uno de los principales factores que se debe considerar al diseñar los dsRNA son las propias dianas moleculares. Entre las dianas se han considerado desde las más convencionales; por ejemplo, genes que codifican enzimas que sintetizan intermediarios en la síntesis del lanosterol (Citocromo P450 lanosterol C-14α-demetilasa, CYP51) (Koch et al., 2016), silenciamiento de efectores (Sarkar y Roy-Barman, 2021) e incluso de los propios componentes del silenciamiento (Wang et al., 2016) (Cuadro 2). Tanto en experimentos realizados in vitro, como en las aplicaciones sobre la superficie de las hojas, se ha observado que el efecto del silenciamiento incrementa al aumentar la dosis de los dsRNA (Haile et al., 2021, McLoughlin et al., 2018, Wang et al., 2016); sin embargo, si la concentración es demasiado alta no se obtienen mejores resultados, lo que sugiere que la maquinaria de iRNA se satura y deja de procesar los dsRNA (McLoughlin et al., 2018). En consecuencia, para lograr los mejores resultados con el SIGS, es necesario caracterizar cada patosistema y determinar la longitud y la concentración más adecuada de dsRNA, para conseguir el control de la enfermedad.
Cuadro 2 Ejemplos exitosos del SIGSy en la inhibición de hongos fitopatógenos.
| Hospedante | Patógeno | Gen diana | Función | Longitud dsRNA (nt) | Máxima persistencia en la planta o in vitro | Referencias |
|---|---|---|---|---|---|---|
| Cebada (Hordeum vulgare) | Fusarium graminearum | FgCYP51A, FgCYP51B, FgCYP51C | Enzima clave en la biosíntesis de Ergosterol | 791 (20 ng µL-1) (CYP51A/CYP51B/CYP51C) | 8 días (6 dpi) z | Koch et al., 2016 |
| Cebada (H. vulgare) | F. graminearum | FgCYP51A, FgCYP51B, FgCYP51C | Enzima clave en la biosíntesis de Ergosterol | 294 (CYP1A), 220 (CYP1B), 238 (CYP1C), 514 (CYP1A/CYP1B), 532 (CYP1A/CYP51C), 458 (CYP1B/CYP51C) | 7 días (5 dpi) z | Koch et al., 2019 |
| Cebada (H. vulgare) | F. graminearum | DCL, AGO | Maquinaria de silenciamiento | 372 (AGO2/DCL1), 1782 (DCL1/DCL2), 1529 (AGO1/AGO2), 1741 (AGO2/DCL2) | 7 días z | Werner et al., 2020 |
| Cebada (H. vulgare) | F. graminearum | FgCYP51A, FgCYP51B, FgCYP51C | Enzima clave en la biosíntesis de Ergosterol | 500 (CYP51A), 400 (CYP51B), 400 (CYP51C), 800 (CYP51A), 800 (CYP51B), 800 (CYP51C), secuencia completa para CYP51A, B y C | 7 días z | Höfle et al., 2020 |
| Jitomate (Solanum lycopersicum) | Botrytis cinerea | DCL1, DCL2 | Maquinaria de silenciamiento | 7 días | Qiao et al., 2021 | |
| Vid (Vitis vinifera) | B. cinerea | BcCYP51, BcChs1, BcEF2 | Biosíntesis de ergosterol, quitina y factor de elongación | 732 (CYP51/Chs1/EF2) (300 µg en 3 mL, absorción por peciolo) | 7 días | Nerva et al., 2020 |
| Vid (V. vinifera) | Plasmopara viticola | DCL | Maquinaria de silenciamiento | 258 (DCL1), 257 (DCL2), 515 (DCL1/DCL2) (75, 100 o 125 ng µL-1) | 14 días | Haile et al., 2021 |
| Arroz (Oryza sativa) | Magnaporthe orizae | MoDES1 | Efector (patogenicidad) | 300 (MoDES1) 300 nM | 10 días z | Sarkar y Roy-Barman, 2021 |
| Fresa (Fragaria vasca) en post cosecha | Botryotinia fuckeliana | DCL, quitina sintasa clase III | Maquinaria de silenciamiento, estructura de la pared celular | dsRNA encapsulado en células de E. coli | 12 días | Islam et al., 2021 |
| Vid (V. vinifera ) en post cosecha | B. cinerea | BcCYP51, BcChs1, BcEF2 | Biosíntesis de ergosterol, quitina y factor de elongación | 732 (CYP51/Chs1/EF2) | 3 días | Nerva et al., 2020 |
| Sin hospedante. Cultivo del hongo en medio sólido (in vitro) | Macrophomina phaseolina | β-1,3-Glucano sintasa (GLS) | Enzima clave en la síntesis de β-1,3 glucano, componente de la pared celular | siRNA (100 nM) | 2.5 días | Forster y Shuai, 2020 |
| Sin hospedante. Cultivo del hongo en medio líquido (in vitro) | F. culmorum | CYP51A, CYP51B, CYP51C | Biosíntesis de Ergosterol | 791 (20 ng µL-1) | 2 días | Koch et al., 2018 |
y Inducción de silenciamiento génico mediante dsRNA asperjados.
z Se consideraron las 48 h previas de tratamiento con dsRNA, más los días de infección monitoreados.
Limitaciones actuales del SIGS en la agricultura. En el sistema SIGS el éxito depende de que los sRNA puedan permeabilizar al interior del tejido del hospedante, lo cual depende de la presencia o ausencia de estomas, del grosor de la cutícula, y del grado de suberización de la epidermis; también depende de la eficiencia de absorción y del procesamiento de los dsRNA por parte del patógeno (Hoffle et al., 2020). Por lo tanto, SIGS es efectivo sobre patógenos que tienen una eficiente absorción de las dsRNA y que poseen una maquinaria funcional de procesamiento de los sRNA. Aunque se trata de un proceso altamente conservado y presente en la mayoría de los organismos, no todos los hongos poseen maquinaria de silenciamiento, por ejemplo, el basidiomiceto Ustilago maydis. Asimismo, parece que el iRNA es poco eficiente para controlar a Zymoseptoria tritici, uno de los hongos más devastadores en los cultivos de trigo (T. aestivum). Kettles y colaboradores (2019) intentaron mediante HIGS silenciar cuatro genes claves de Z. tritici, pero ninguno fue exitoso, demostrando la incapacidad del hongo de absorber dsRNA a través de la interacción con trigo. Posteriormente, Ma y colaboradores (2020) reportaron que durante la colonización de trigo por Z. tritici no identificaron la presencia de iRNA, por lo que concluyeron que el mecanismo de silenciamiento no participa en esta infección. A pesar de que Z. tritici mantiene los componentes del iRNA, parece no tener un papel durante la infección del hospedante. Esto sugiere que para el éxito de SIGS es clave que exista una intercomunicación natural entre patógeno y hospedante por medio de sRNA. Recientemente Qiao y colaboradores (2021) realizaron un ensayo en el que usaron un dsRNA fluorescente para evaluar la capacidad de absorción de varios hongos in vitro. Encontraron que Colletotrichum gloeosporioides es incapaz de absorberlos, mientras que en B. cinerea, V. dahliae, Rhizoctonia solani, Aspergillus niger y S. sclerotiorum la fluorescencia se observó en el interior de sus células a las 6 h, lo que indica la internalización de los dsRNAs. Otros microorganismos como Trichoderma virens y Phytophthora infestans absorbieron los dsRNA de manera limitada. Curiosamente, Mahto y colaboradores (2020) lograron silenciar un gen de C. gloeosporioides y controlar la antracnosis en chile (Capsicum annuum) y jitomate donde emplearon HIGS y propusieron que los sRNA atraviesan el haustorio del hongo a través de vesículas extracelulares secretadas por la planta. Sin embargo, es importante recordar que el silenciamiento mediante HIGS no funcionó en Z. tritici (Kettles et al., 2019), lo que sugiere que los métodos de ingreso de los iRNA pueden variar entre los microrganismos y en algunos casos no existir. Para la mayoría de los hongos fitopatógenos se desconocen sus capacidades de absorción y procesamiento de los dsRNAs, por lo que se requiere ampliar estas pruebas para establecer mejor el potencial de SIGS para el control de hongos fitopatógenos.
Potencial del ARN de interferencia en cultivos agrícolas de méxico y américa latina
América Latina y el Caribe (ALC) es una importante región productora de alimentos en el mundo. Sus principales productos agrícolas son cereales, semillas oleaginosas, banano, café, azúcar, frutas y verduras. Adicionalmente, 26% de la producción global de los principales frutos tropicales [bananas (M. acuminata), mango (Mangifera indica), piña (Ananas comosus), aguacate (Persea americana) y papaya (Carica papaya)] se cultivan en ALC, principalmente en Brasil, Ecuador, México y Costa Rica. ALC es una importante región exportadora y clave en la economía global. Por ejemplo, los bananos y los plátanos (M. balbisiana) se encuentran entre los productos alimenticios más importantes en el mundo y su comercialización es un pilar importante en la economía de muchos países de ALC. Sin embargo, su producción es constantemente amenazada por diversas enfermedades, entre las que destacan la Sigatoka negra (SN) cuyo agente causal es el hongo ascomiceto Pseudocercospora fijiensis, y el marchitamiento por Fusarium, también llamado Mal de panamá. La SN es una enfermedad foliar que reduce significativamente la tasa de fotosíntesis en la planta, provocando su debilitamiento y eventualmente su muerte (Chí-Manzanero et al., 2021). Por otro lado, el Mal de Panamá provocado por Fusarium oxysporum f. sp. cubense raza 1 (Foc1), acabó con el cultivo del banano Gros Michel, obligando a los productores a cambiar a un cultivo más resistente (cultivar Cavendish). Sin embargo, con la aparición de Fusarium oxysporum f. sp. cubense raza 4 (Foc4), la industria bananera corre un grave peligro. La urgente necesidad de controlar esta peligrosa raza de Foc ha llevado a evaluar la tecnología del silenciamiento por iRNA mediante HIGS (Ghag y Ganapathi, 2019). Estos autores desarrollaron líneas transgénicas de banano que expresan iRNA contra el gen velvet y el Factor 1 de la transcripción de Foc4, ambos genes relacionados con el crecimiento, desarrollo y la patogénesis; estas plantas sobrevivieron seis semanas y algunas hasta ocho meses sin generar síntomas (Ghag et al., 2014). Así mismo, se han generado líneas transgénicas de banano capaces de silenciar los transcritos de la enzima C-24 esterol metiltransferasa (ERG6) y del citocromo P450 lanosterol C-14α dimetilasa (ERG11), ambas enzimas involucradas en la síntesis del ergoesterol; ambas líneas fueron evaluadas hasta por dos años (Dou et al., 2020).
En otros cultivos, se estima que la producción global de mango y aguacate seguirá creciendo en ALC. Brasil y México son los mayores productores de mango y aguacate, y de este último, México es el mayor exportador, mientras que Colombia, República Dominicana y Perú han aumentado su producción; ALC cubre el 73% de la producción global de aguacate en el mundo, lo que resalta su importancia en la agricultura de la región. Entre estos frutos tropicales, la enfermedad antracnosis (Colletotrichum sp.), es común en condiciones de post-cosecha. En mango y aguacate, la antracnosis es causada por C. gloeosporioides y este hongo ataca al fruto en pre- y post-cosecha, y también causa daño foliar. La evidencia disponible en la literatura sugiere que C. gloeosporioides no absorbe eficientemente los dsRNA exógenos (Qiao et al., 2021). Sin embargo, se ha demostrado que la antracnosis en chile y jitomate puede ser controlado por medio de HIGS (Mahto et al., 2020), lo que sugiere que el uso de vesículas como acarreadores pudiera ser una alternativa para el control por SIGS. Entre las plagas que atacan cultivos de importancia en ALC destaca el gusano cogollero (Spodoptera frugiperda), una plaga polífaga que daña diferentes cultivos vegetales, preferentemente a maíz, sorgo (Sorghum bicolor), algodón y soya, cebolla, zanahoria (Daucus carota), lechuga, papaya, sandía (Citrullus lanatus), melón (Cucumis melo), pepino (Cucumis sativus), aguacate, banana, arroz, café (Coffea arabica), jitomate, cacao, caña de azúcar, entre muchos otros (Montezano et al., 2018). Esta plaga se encuentra ampliamente distribuida en ALC y parte de Norte América y sus larvas pueden ocasionar pérdidas de hasta el 100% en los cultivos. Recientemente Gurusamy y colaboradores (2020a; 2020b) describieron el control de S. frugiperda mediante iRNA, empleando formulaciones de dsRNA con lípidos catiónicos que protegen los dsRNA de la degradación en el interior del insecto y eficientizan el silenciamiento (Gurusamy et al., 2020a). En otro reporte, los mismos investigadores evaluaron el uso de nanopartículas para la protección de los dsRNA, ponderando las formulaciones con quitosano por su capacidad de protección y por ser un polímero biodegradable, no tóxico, económico y seguro con el ambiente, demostrando una mejora en la eficiencia del silenciamiento por SIGS (Gurusamy et al., 2020b). Otra plaga de gran importancia en ALC es la broca de café (Hypothenemus hampei). Este insecto es responsable de las mayores pérdidas en el cultivo de café en el mundo (Jaramillo et al., 2006). La aplicación de fragmentos de dsRNA en la cavidad preoral de las larvas de la broca es efectiva para el silenciamiento génico, al igual que la aplicación exógena para la adquisición por ingestión (Aguilera et al., 2011). Entre los genes diana evaluados, los mejores resultados fueron obtenidos silenciando las mananasas o xilanasas involucradas en la hidrólisis de los principales carbohidratos del grano de café (Aguilera et al., 2011). La mosquita blanca (Bemicia tabaci) es otro insecto plaga importante, ampliamente distribuida por toda ALC. Este insecto afecta cultivos de aguacate, frijol (Phaseolus vulgaris), jitomate, chile y otras hortalizas; al alimentarse del floema de las plantas, provocando desórdenes fisiológicos. Adicionalmente, es vector de Begomovirus. A la fecha se ha reportado el silenciamiento de numerosos genes diana de B. tabaci a través de HIGS o de dietas artificial que contenga dsRNA (Grover et al., 2019), demostrando que la tecnología del iRNA es una alternativa prometedora para su control. Sin embargo, aún no hay reportes de la evaluación en campo del control de B. tabaci con alguna de estas tecnologías, aunque recientemente se ha demostrado el potencial de control mediante SIGS, en combinación con nanopartículas de doble hidróxido en capas de MgFe (MgFe-LDH “Bioclay”) (Jain et al., 2022). Así mismo se ha reportado el potencial de secuencias homólogas derivadas del virus Pepper golden mosaic virus (PepGMV) y heterólogas derivadas de Tomato chino La Paz virus (ToChLPV), abriendo las posibilidad de protección contra Pepper golden mosaic virus (PepGMV) (Medina-Hernández et al., 2013; Vargas-Salinas et al., 2021). A pesar de ser promisorios para un manejo integrado y amigable para el medio ambiente y humano, aún se desconoce el potencial de los dsRNAs y en particular de SIGS en la agricultura de ALC.
En México, se han empleado iRNA en el uso de VIGS para estudiar genes vegetales. Álvarez-Venegas et al. (2011) silenciaron genes relacionados con la floración en canola (B. napus) y obtuvieron plantas que florecen sin vernalización. En otro trabajo, Villanueva-Hernández et al. (2013), crearon un vector VIGS empleando un Begomovirus aislado en Yucatán en plantas del género Euphorbia, al que le llamaron pEuMV-YP y con el que silenciaron ChlI como gen reportero para evaluar la funcionalidad del vector VIGS; ChlI codifica una proteína que protege la clorofila, y su silenciamiento genera un fenotipo de fotoblanqueo. Los autores demostraron que pEuMV-YP es adecuado para silenciar genes en plantas de N. benthamiana y de C. annuum. Similarmente, Arce-Rodríguez y Ochoa-Alejo (2020), crearon un vector viral basado en el virus del sonajero del tabaco (Tobacco rattle virus, TRV), al que le incorporaron un casete de tecnología Gateway para clonar en él, el ADN mediante recombinación, y también le incorporaron la región T-ADN de A. tumefasciens para introducir el constructo VIGS mediante agroinfiltración. Como prueba de concepto se utilizó este vector VIGS para silenciar el gen que codifica la fitoeno desaturasa en chile, lo que disminuyó la síntesis de fitoeno, un precursor de los carotenos, y generó un fenotipo de fotoblanqueo en las hojas. Recientemente, Villanueva- Hernández et al. (2022), usaron el pEuMV-YP para expresar en N. benthamiana un siRNA contra el gen Krt18 de ratón (Mus musculus), demostrando que la expresión en planta mediada por VIGS puede ser usada para silenciar genes en organismos de diferentes reinos, en este caso, de plantas a mamíferos. De manera natural, consumimos ARNs pequeños de algunas plantas que usamos como alimentos o medicinas. Por ejemplo, en China, se usa la planta Lonicera japonica para combatir la influenza A y el virus SARS-CoV-2 y se ha demostrado que su principio activo es un ARN pequeño de tipo miRNA, el miRNA2911 (Zhou et al., 2015; 2020), por lo que la producción dirigida de dsRNA en plantas contra patógenos de humanos puede ser de interés médico, aunque todavía quedan muchos puntos por resolver como la baja estabilidad de los sRNAs y alcanzar una concentración suficiente que sea terapéutica. Aunque aún es limitado el número de trabajos de silenciamiento en plantas o fitopatógenos en México, las herramientas metodológicas que se han ido construyendo permiten esperar que las investigaciones en este campo vayan en aumento.
Desafíos para establecer sigs en el campo
Se ha evaluado el potencial de los iRNA en los cultivos tropicales utilizando la estrategia de generación de plantas transgénicas (HIGS); aunque los resultados han sido prometedores, se tiene limitaciones en las legislaciones de la mayoría de los países en ALC para el uso agronómico de materiales genéticamente modificados. Sin embargo, los resultados han revelado el potencial del iRNA para controlar por ejemplo, a Foc4, y abren camino para evaluar la alternativa no transgénica, el SIGS (Dou et al., 2020; Ghag et al., 2014; Koch et al., 2019; Koch y Wassenegger, 2021; Mahto et al., 2020; Montezano et al., 2018; Qi et al., 2018; Qiao et al., 2021; Zhu et al., 2017).
Para conseguir una exitosa protección de plantas y frutos en post-cosecha de interés agronómico mediante el uso de SIGS, se requiere estudiar la funcionalidad de los dsRNA en la interacción planta-patógeno. Uno de los desafíos es construir nanovehículos que protejan y alarguen la estabilidad de los dsRNA, y que sean inocuos para el hombre y demás seres vivos, además de ser biodegradables. Una nanopartícula que cumple estas características es una arcilla de doble hidróxido que forma nanoláminas en capas (llamada “bioclay”), que nanoencapsula los dsRNA y los mantiene estables hasta 20 días (Gebremichael et al., 2021). Fue inicialmente desarrollada para conferir protección contra virus fitopatógenos, pero actualmente se está evaluando para protección vegetal contra enfermedades fúngicas. Otro reto por superar es mejorar la absorción de los dsRNA en los hongos que sí poseen la maquinaria de silenciamiento, pero que resultan recalcitrantes, como C. glosporoides. Así como obtener costos más rentables de producción de los dsRNAs. Las próximas investigaciones deberán enfocarse en resolver estos retos para llevar a SIGS de promesa a realidad. También se requiere trabajar en la normatividad, para que eventualmente pueda llegar a ser una tecnología comercial.
Perspectivas de la sigs
Es importante enfocar los esfuerzos para superar las limitantes técnicas de SIGS, pues su perspectiva es enorme. La selección de posibles dianas es muy amplia; prácticamente cualquier gen involucrado en la viabilidad o en la patogenicidad de un organismo es susceptible de ser diana, y la facilidad de producción de los dsRNA hace que esta tecnología sea amigable para estudios en alta escala. Sus aplicaciones comerciales más cercanas son el control de insectos vectores de enfermedades de plantas y humanos (Castellanos et al., 2019; Zheng et al., 2019; Zotti et al., 2018), el control de enfermedades de frutos post-cosecha (de Oliveira et al., 2021; Qiao et al., 2021) y el control de malezas (Zabala-Pardo et al., 2022), porque ya existen reportes que evidencian bajo qué condiciones pueden ser efectivos en estos casos.
Conclusiones
La tecnología del iRNA tiene gran potencial para el control de plagas y enfermedades causadas por hongos, en particular la estrategia SIGS, cuya aplicación es segura y ecoamigable. Es crucial desarrollar investigaciones en esta dirección sobre los patosistemas de importancia agronómica en México y Latinoamérica, para crear un catálogo de los patógenos que pueden ser controlados por SIGS.
Agradecimientos
Este trabajo fue financiado por el Consejo Nacional de Humanidades, Ciencias y Tecnologías (CONAHCyT), con los Proyectos FOSEC-SEP 220957, y FOP16-2021-01 No. 320993, y la beca #644399 a OJCD y #700673 a KGCA.
REFERENCIAS
Aguilera GC, Padilla E, Flórez CP, Rubio JD y Acuña JR. 2011. ARN interferente: Potenciales usos en genómica funcional y control genético de Hypothenemus hampei (coleoptera: Scolytinae). Revista Colombiana de Entomologia 37(2): 167-172. http://www.scielo.org.co/scielo.php?pid=S0120-04882011000200001&script=sci_arttext&tlng=es [ Links ]
Álvarez-Venegas R, Zhang Y, Kraling Konrad and Tulsieram L. 2011. Flowering without vernalization in winter canola (Brassica napus): use of Virus-Induced Gene Silencing (VIGS) to accelerate genetic gain. Nova scientia 3(5): 29-50. http://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S2007-07052011000100003&lng=es&tlng=en [ Links ]
Arce-Rodríguez ML and Ochoa-Alejo N. 2020. Virus-Induced Gene Silencing (VIGS) in Chili Pepper (Capsicum spp.). Methods in molecular biology (Clifton, N.J.), 2172: 27-38. https://doi.org/10.1007/978-1-0716-0751-0_3 [ Links ]
Brito FSD, Santos JRP, Azevedo VCR, Peixouto YS, de Oliveira SA, Ferreira CF, Haddad F, Amorim EP, Fraaije B and Miller RNG. 2020. Genetic diversity and azole fungicide sensitivity in Pseudocercospora musae field populations in Brazil. Frontiers in Microbiology 11(99): 1-13. https://doi.org/10.3389/fmicb.2020.00099 [ Links ]
Cai Q, He B, Kogel KH and Jin H. 2018. Cross-kingdom RNA trafficking and environmental RNAi-nature’s blueprint for modern crop protection strategies. Current opinion in microbiology 46: 58-64. https://doi.org/10.1016/j.mib.2018.02.003 [ Links ]
Castellanos NL, Smagghe G, Sharma R, Oliveira EE and Christiaens O. 2019. Liposome encapsulation and EDTA formulation of dsRNA targeting essential genes increase oral RNAi-caused mortality in the Neotropical stink bug Euschistus heros. Pest. Managment Science 75: 537-548. doi: 10.1002/ps.5167 [ Links ]
Chí-Manzanero B, Carreón-Anguiano KG, Todd JNA, Góme- Tah R, Grijalva-Arango R, Tzec- Simá MA and Canto-Canché B. 2021. Analysis of Pseudocercospora fijiensis genes upregulated during early interaction with Musa acuminata (var. dwarf cavendish). Bionatura 6(1): 1540-1546. https://doi.org/10.21931/RB/2021.06.01.15 [ Links ]
Dalakouras A, Jarausch W, Buchholz G, Bassler A, Braun M, Manthey T, Krczal G and Wassenegger M. 2018. Delivery of hairpin rnas and small RNAs into woody and herbaceous plants by trunk injection and petiole absorption. Frontiers in Plant Science 9: 1-11. https://doi.org/10.3389/fpls.2018.01253 [ Links ]
Dalakouras A, Wassenegger M, Dadami E, Ganopoulos I, Pappas ML and Papadopoulou K. 2020. Genetically modified organism-free RNA interference: Exogenous application of RNA molecules in plants. Plant Physiology 182: 1-13. https://doi.org/10.1104/pp.19.00570 [ Links ]
de Oliveira-Filho JG, Silva GDC, Cipriano L, Gomes M and Egea MB. 2021. Control of postharvest fungal diseases in fruits using external application of RNAi. Journal of Food Science 86(8): 3341-3348. https://doi.org/10.1111/1750-3841.15816 [ Links ]
Dou T, Shao X, Hu C, Liu S, Sheng O, Bi F, Deng G, Ding L, Li C, Dong T, Gao H, He W, Peng X, Zhang S, Huo H, Yang Q and Yi G. 2020. Host-induced gene silencing of Foc TR4 ERG6/11 genes exhibits superior resistance to Fusarium wilt of banana. Plant Biotechnology Journal 18: 11-13. https://doi.org/10.1111/pbi.13204 [ Links ]
Fletcher SJ, Reeves PT, Hoang BT and Mitter N. 2020. A perspective on RNAi-based biopesticides. Frontiers in Plant Science 11: 51. https://doi.org/10.3389/fpls.2020.00051 [ Links ]
Forster H and Shuai B. 2020. RNAi-mediated knockdown of β-1,3-glucan synthase suppresses growth of the phytopathogenic fungus Macrophomina phaseolina. Physiological and Molecular Plant Pathology 110: 101486. https://doi.org/10.1016/j.pmpp.2020.101486 [ Links ]
Gaffar FY, Imani J, Karlovsky P, Koch A and Kogel KH. 2019. Different components of the RNA interference machinery are required for conidiation, ascosporogenesis, virulence, deoxynivalenol production, and fungal inhibition by exogenous double-stranded rna in the head blight pathogen Fusarium graminearum. Frontiers in Microbiology 10: 1662. https://doi.org/10.3389/fmicb.2019.01662 [ Links ]
Garcia-Ruiz H, Ruiz M, Peralta S, Gabriel C and El-Mounadi K. 2016. Mechanisms, applications, and perspectives of antiviral RNA silencing in plants. Revista Mexicana de Fitopatologia 34(3): 286-307. https://doi.org/10.18781/R.MEX.FIT.1606-8 [ Links ]
Ghag SB and Ganapathi TR. 2019. RNAi-mediated protection against banana diseases and pests. 3 Biotech 9(3): 112. https://doi.org/10.1007/s13205-019-1650-7 [ Links ]
Ghag SB, Shekhawat UKS and Ganapathi TR. 2014. Host-induced post-transcriptional hairpin RNA-mediated gene silencing of vital fungal genes confers efficient resistance against Fusarium wilt in banana. Plant Biotechnology Journal 12: 541-553. https://doi.org/10.1111/pbi.12158 [ Links ]
Grover S, Jindal V, Banta G, Taning CNT, Smagghe G and Christiaens O. 2019. Potential of RNA interference in the study and management of the whitefly, Bemisia tabaci. Archives of Insect Biochemistry and Physiology 100(2): 1-17. https://doi.org/10.1002/arch.21522 [ Links ]
Gurusamy D, Mogilicherla K and Palli SR. 2020. Chitosan nanoparticles help double-stranded RNA escape from endosomes and improve RNA interference in the fall armyworm, Spodoptera frugiperda. Archives of Insect Biochemistry and Physiology 104(4): 1-15. https://doi.org/10.1002/arch.21677 [ Links ]
Gurusamy D, Mogilicherla K, Shukla JN and Palli SR. 2020. Lipids help double-stranded RNA in endosomal escape and improve RNA interference in the fall armyworm, Spodoptera frugiperda. Archives of Insect Biochemistry and Physiology 104(4): 1-19. https://doi.org/10.1002/arch.21678 [ Links ]
Haile ZM, Gebremichael DE, Capriotti L, Molesini B, Negrini F, Collina M, Sabbadini S, Mezzetti B and Baraldi E. 2021. Double-stranded RNA targeting dicer-like genes compromises the pathogenicity of Plasmopara viticola on grapevine. Frontiers in Plant Science 12: 1-7. https://doi.org/10.3389/fpls.2021.667539 [ Links ]
Halder K, Chaudhuri A, Abdin MZ, Majee M and Datta A. 2022. RNA interference for improving disease resistance in plants and its relevance in this clustered regularly interspaced short palindromic repeats-dominated era in terms of dsrna-based biopesticides. Frontiers in plant science 13: 885128. https://doi.org/10.3389/fpls.2022.885128 [ Links ]
Höfle L, Biedenkopf D, Werner B. T, Shrestha A, Jelonek L and Koch A. 2020. Study on the efficiency of dsRNAs with increasing length in RNA-based silencing of the Fusarium CYP51 genes. RNA Biology 17(4): 463-473. https://doi.org/10.1080/15476286.2019.1700033 [ Links ]
Hudzik C, Hou Y, Ma W and Axtell MJ. 2020. Exchange of small regulatory RNAs between plants and their pests. Plant Physiology 182: 51-62. https://doi.org/10.1104/pp.19.00931 [ Links ]
Islam MT, Davis Z, Chen L, Englaender J, Zomorodi S, Frank J, Bartlett K, Somers E, Carballo SM, Kester M, Shakeel A, Pourtaheri P and Sherif SM. 2021. Minicell-based fungal RNAi delivery for sustainable crop protection. Microbial Biotechnology 14(4): 1847-1856. https://doi.org/10.1111/1751-7915.13699 [ Links ]
Jain RG, Fletcher SJ, Manzie N, Robinson KE, Li P, Lu E, Brosnan CA, Xu ZP and Mitter N. 2022. Foliar application of clay-delivered RNA interference for whitefly control. Nature Plants 8(5): 535-548. https://doi.org/10.1038/s41477-022-01152-8 [ Links ]
Jaramillo J, Borgemeister C and Baker P. 2006. Coffee berry borer Hypothenemus hampei (Coleoptera: Curculionidae): searching for sustainable control strategies. Bulletin of Entomological Research 96(3): 223-233. https://doi.org/10.1079/ber2006434 [ Links ]
Jensen PD, Zhang Y, Wiggins BE, Petrick JS, Zhu J, Kerstetter RA, Heck GR and Ivashuta SI. 2013. Computational sequence analysis of predicted long dsRNA transcriptomes of major crops reveals sequence complementarity with human genes. GM Crops and Food 4(2): 90-97. https://doi.org/10.4161/gmcr.25285 [ Links ]
Jian J and Liang X. 2019. One small RNA of Fusarium graminearum targets and silences CEBiP gene in common wheat. Microorganisms 7(10): 425. https://doi.org/10.3390/microorganisms7100425 [ Links ]
Jin Y, Zhao JH, Zhao P, Zhang T, Wang Sand Guo HS. 2019. A fungal milRNA mediates epigenetic repression of a virulence gene in Verticillium dahliae. Philosophical transactions of the Royal Society of London 374(1767): 20180309. https://doi.org/10.1098/rstb.2018.0309 [ Links ]
Kaldis A, Berbati M, Melita O, Reppa C, Holeva M, Otten P and Voloudakis A. 2018. Exogenously applied dsRNA molecules deriving from the Zucchini yellow mosaic virus (ZYMV) genome move systemically and protect cucurbits against ZYMV. Molecular Plant Pathology 19(4): 883-895. https://doi.org/10.1111/mpp.12572 [ Links ]
Kettles GJ, Hofinger BJ, Hu P, Bayon C, Rudd JJ, Balmer D, Courbot M, Hammond-Kosack K E, Scalliet G and Kanyuka K. 2019. sRNA profiling combined with gene function analysis reveals a lack of evidence for cross-kingdom RNAi in the wheat - Zymoseptoria tritici pathosystem. Frontiers in Plant Science 10: 892. https://doi.org/10.3389/fpls.2019.00892 [ Links ]
Kiselev KV, Suprun AR, Aleynova OA, Ogneva ZV and Dubrovina AS. 2021. Physiological conditions and dsRNA application approaches for exogenously induced RNA interference in Arabidopsis thaliana. Plants 10(264): 1-13. https://doi.org/10.3390/plants10020264 [ Links ]
Koch A, Biedenkopf D, Furch A, Weber L, Rossbach O, Abdellatef E, Linicus L, Johannsmeier J, Jelonek L, Goesmann A, Cardoza V, McMillan J, Mentzel T and Kogel KH. 2016. An RNAi-based control of Fusarium graminearum infections through spraying of long dsRNAs involves a plant passage and is controlled by the fungal silencing machinery. PLoS Pathogens 12(10): 1-22. https://doi.org/10.1371/journal.ppat.1005901 [ Links ]
Koch A, Höfle L, Werner BT, Imani J, Schmidt A, Jelonek L and Kogel KH. 2019. SIGS vs HIGS: a study on the efficacy of two dsRNA delivery strategies to silence Fusarium FgCYP51 genes in infected host and non-host plants. Molecular Plant Pathology 20(12): 1636-1644. https://doi.org/10.1111/mpp.12866 [ Links ]
Koch A, Stein E and Kogel KH. 2018. RNA-based disease control as a complementary measure to fight Fusarium fungi through silencing of the azole target Cytochrome P450 Lanosterol C-14 α-Demethylase. European Journal of Plant Pathology 152(4): 1003-1010. https://doi.org/10.1007/s10658-018-1518-4 [ Links ]
Koch A and Wassenegger M. 2021. Host-induced gene silencing - mechanisms and applications. New Phytologist 231(1): 54-59. https://doi.org/10.1111/nph.17364 [ Links ]
Lax C, Tahiri G, Patiño-Medina JA, Cánovas-Márquez JT, Pérez-Ruiz JA, Osorio-Concepción M, Navarro E and Calo S. 2020. The evolutionary significance of RNAi in the fungal kingdom. International Journal of Molecular Science 21(24): 9348. https://doi.org/10.3390/ijms21249348. PMID: 33302447; PMCID: PMC7763443 [ Links ]
Li C and Zamore PD. 2019. Preparation of dsRNAs for RNAi by in vitro transcription. Cold Spring Harbor protocols (3). https://doi.org/10.1101/pdb.prot097469 [ Links ]
Ma X, Wiedmer J and Palma-Guerrero J. 2020. Interaction between wheat and Zymoseptoria tritici. Small RNA bidirectional crosstalk during the interaction between wheat and Zymoseptoria tritici. Frontiers in Plant Science 10: 1669. https://doi.org/10.3389/fpls.2019.01669 [ Links ]
Mahto BK, Singh A, Pareek M, Rajam MV, Dhar-Ray S and Reddy PM. 2020. Host-induced silencing of the Colletotrichum gloeosporioides conidial morphology 1 gene (CgCOM1) confers resistance against Anthracnose disease in chilli and tomato. Plant Molecular Biology 104: 381-395. https://doi.org/10.1007/s11103-020-01046-3 [ Links ]
Majumdar R, Rajasekaran K and Cary JW. 2017. RNA Interference (RNAi) as a potential tool for control of mycotoxin contamination in crop plants: concepts and considerations. Frontiers in Plant Science 8(200): 1-14. https://doi.org/10.3389/fpls.2017.00200 [ Links ]
McLoughlin AG, Wytinck N, Walker PL, Girard IJ, Rashid KY, De Kievit T, Fernando WGD, Whyard S and Belmonte MF. 2018. Identification and application of exogenous dsRNA confers plant protection against Sclerotinia sclerotiorum and Botrytis cinerea. Scientific Reports 8: 7320. https://doi.org/10.1038/s41598-018-25434-4 [ Links ]
Medina-Hernández D, Rivera-Bustamante RF, Tenllado F and Holguín-Peña RJ. 2013. Effects and effectiveness of two RNAi constructs for resistance to pepper golden mosaic virus in Nicotiana benthamiana plants. Viruses 5(12): 2931-2945. https://doi.org/10.3390/v5122931 [ Links ]
Montezano DG, Specht A, Sosa-Gómez DR, Roque-Specht VF, Sousa-Silva JC, Paula-Moraes SV, Peterson JA and Hunt TE. 2018. Host Plants of Spodoptera frugiperda (Lepidoptera : Noctuidae) in the Americas. African Entomology 26(2): 286-300. https://doi.org/doi.org/10.4001/003.026.0286 [ Links ]
Nerva L, Sandrini M, Gambino G and Chitarra W. 2020. Double-stranded RNAs (dsRNAs) as a sustainable tool against gray mold (Botrytis cinerea) in grapevine: effectiveness of different application methods in an open-air environment. Biomolecules 10(2): 200. https://doi.org/10.3390/biom10020200 [ Links ]
Papadopoulou N, Devos Y, Álvarez-Alfageme F, Lanzoni A and Waigmann E. 2020. Risk assessment considerations for genetically modified RNAi plants: EFSA’s activities and perspective. Frontiers in Plant Science 11: 445. https://doi.org/10.3389/fpls.2020.00445 [ Links ]
Parker JE, Warrilow AGS, Price CL, Mullins JGL, Kelly DE and Kelly SL. 2014. Resistance to antifungals that target CYP51. Journal of Chemical Biology 7(4): 143-161. https://doi.org/10.1007/s12154-014-0121-1 [ Links ]
Qi T, Guo J, Peng H, Liu P, Kang Z and Guo J. 2019. Host-induced gene silencing: a powerful strategy to control diseases of wheat and barley. International Journal of Molecular Sciences 20(206): 1-15. https://doi.org/10.3390/ijms20010206 [ Links ]
Qi T, Zhu X, Tan C, Liu P, Guo J, Kang Z and Guo J. 2018. Host-induced gene silencing of an important pathogenicity factor PsCPK1 in Puccinia striiformis f. sp. tritici enhances resistance of wheat to stripe rust. Plant Biotechnology Journal 16(3): 797-807. https://doi.org/10.1111/pbi.12829 [ Links ]
Qiao L, Lan C, Capriotti L, Ah-Fong A, Nino Sanchez J, Hamby R, Heller J, Zhao H, Glass NL, Judelson HS, Mezzetti B, Niu D and Jin H. 2021. Spray-induced gene silencing for disease control is dependent on the efficiency of pathogen RNA uptake. Plant Biotechnology Journal 19(9): 1756-1768. https://doi.org/10.1111/pbi.13589 [ Links ]
Rodrigues TB and Petrick JS. 2020. Safety considerations for humans and other vertebrates regarding agricultural uses of externally applied RNA molecules. Frontiers in Plant Science 11: 407. https://doi.org/10.3389/fpls.2020.00407 [ Links ]
Rosa C, Kuo Y, Wuriyanghan H and Falk BW. 2018. RNA interference mechanisms and applications in plant pathology. Annual Review of Phytopatology 56. 581-610. [ Links ]
Sang H and Kim JI. 2020. Advanced strategies to control plant pathogenic fungi by host-induced gene silencing (HIGS) and spray-induced gene silencing (SIGS). Plant Biotechnology Reports 14: 1-8. https://doi.org/10.1007/s11816-019-00588-3 [ Links ]
Sarkar A and Roy-Barman S. 2021. Spray-induced silencing of pathogenicity gene MoDES1 via exogenous double-stranded RNA can confer partial resistance against fungal blast in rice. Frontiers in Plant Science 12: 1-19. https://doi.org/10.3389/fpls.2021.733129 [ Links ]
Savary S, Willocquet L, Pethybridge SJ, Esker P, McRoberts N and Nelson A. 2019. The global burden of pathogens and pests on major food crops. Nature Ecology and Evolution 3(3): 430-439. https://doi.org/10.1038/s41559-018-0793-y [ Links ]
Sun G and Riggs AD. 2017. A Simple and cost-effective approach for in vitro production of sliced siRNAs as potent triggers for RNAi. Molecular therapy. Nucleic acids 8: 345-355. https://doi.org/10.1016/j.omtn.2017.07.008 [ Links ]
Taning CNT, Arpaia S, Christiaens O, Dietz-Pfeilstetter A, Jones H, Mezzetti B, Sabbadini S, Sorteberg HG, Sweet J, Ventura V and Smagghe G. 2020. RNA-based biocontrol compounds: current status and perspectives to reach the market. Pest Management Science 76(3): 841-845. https://doi.org/10.1002/ps.5686 [ Links ]
Todd JNA, Carreón-Anguiano KG, Couoh-Dzul OJ, de los Santos-Briones C and Blondy Canto-Canché. 2023. Effectors: key actors in phytopathology. Mexican Journal of Phytopathology 41(2): 203-228. https://doi.org/10.18781/R.MEX.FIT.2210-4 [ Links ]
van Dijk M, Morley T, Rau ML and Saghai Y. 2021. A meta-analysis of projected global food demand and population at risk of hunger for the period 2010-2050. Nature Food 2(7): 494-501. https://doi.org/10.1038/s43016-021-00322-9 [ Links ]
Vargas-Salinas M, Medina-Hernández D, Arcos-Ortega GF, Luis-Villaseñor IE and Holguín-Peña RJ. 2021. RNAi activation with homologous and heterologous sequences that induce resistance against the begomovirus Pepper golden mosaic virus (PepGMV). 3 Biotech 11(3): 1-19. https://doi.org/10.1007/s13205-021-02653-7 [ Links ]
Villanueva-Alonzo HJ, Haro-Álvarez AP, Alvarado-Segura AA, Valle-Gough RE, Collí-Mull JG, Cal-Torres A, Arana-Argáez VE, Torres-Romero JC, Moreno-Valenzuela OA, Nic-Can G, Ayil-Gutiérrez BA and Acosta-Viana KY. 2022. A method to produce vsiRNAs in plants with cross-kingdom gene silencing capacity. Applied Sciences 12(11): 5329. https://doi.org/10.3390/app12115329 [ Links ]
Villanueva-Alonzo HJ, Us-Camas RY, López-Ochoa LA, Robertson D, Guerra-Peraza O, Minero-García Y and Moreno-Valenzuela OA. 2013. A new virus-induced gene silencing vector based on Euphorbia mosaic virus-Yucatan peninsula for NPR1 silencing in Nicotiana benthamiana and Capsicum annuum var. Anaheim. Biotechnology letters 35(5): 811-823. https://doi.org/10.1007/s10529-013-1146-1 [ Links ]
Wang M and Jin H. 2017. Spray-induced gene silencing: a powerful innovative strategy for crop protection. Trends in Microbiology 25(1): 4-6. https://doi.org/10.1016/j.tim.2016.11.011 [ Links ]
Wang B, Sun Y, Song N, Zhao M, Liu R, Feng H, Wang X and Kang Z. 2017. Puccinia striiformis f. sp. tritici microRNA-like RNA 1 (Pst-milR1), an important pathogenicity factor of Pst, impairs wheat resistance to Pst by suppressing the wheat pathogenesis-related 2 gene. The New phytologist 215(1): 338-350. https://doi.org/10.1111/nph.14577 [ Links ]
Wang M, Weiberg A, Lin FM, Thomma BPHJ, Huang H and Jin H. 2016. Bidirectional cross-kingdom RNAi and fungal uptake of external RNAs confer plant protection. Nature Plants 2: 1-10. https://doi.org/10.1038/nplants.2016.151 [ Links ]
Weiberg A, Wang M, Lin FM, Zhao H, Zhang Z, Kaloshian I, Huang HD and Jin H. 2013. Fungal small RNAs suppress plant immunity by hijacking host RNA interference pathways. Science 342(6154): 118-123. https://doi.org/10.1126/science.1239705 [ Links ]
Werner BT, Gaffar FY, Schuemann J, Biedenkopf D and Koch AM. 2020. RNA-spray-mediated silencing of Fusarium graminearum AGO and DCL genes improve barley disease resistance. Frontiers in Plant Science 11(476): 1-11. https://doi.org/10.3389/fpls.2020.00476 [ Links ]
Werner BT, Koch A, Šečić E, Engelhardt J, Jelonek L, Steinbrenner J and Kogel KH. 2021. Fusarium graminearum DICER-like-dependent sRNAs are required for the suppression of host immune genes and full virulence. PLoS One16(8):e0252365. https://doi.org/10.1371/journal.pone.0252365. PMID: 34351929; PMCID: PMC8341482 [ Links ]
Yin C and Hulbert S. 2015. Host Induced Gene Silencing (HIGS), a promising strategy for developing disease resistant crops. Gene Technology 4(3): 1-2. https://doi.org/10.4172/2329-6682.1000130 [ Links ]
Yin C, Jurgenson JE and Hulbert SH. 2011. Development of a host-induced RNAi system in the wheat stripe rust fungus Puccinia striiformis f. sp. tritici. Molecular Plant-Microbe Interactions 24(5): 554-561. https://doi.org/10.1094/MPMI-10-10-0229 [ Links ]
Yin C, Zhu H, Jiang Y, Shan Y and Gong L. 2020. Silencing Dicer-like genes reduces virulence and sRNA generation in Penicillium italicum, the cause of citrus blue mold. Cells 9(2): 363. https://doi.org/10.3390/cells9020363 [ Links ]
Xu M, Guo Y, Tian R, Gao C, Guo F, Voegele RT, Bao J, Li C, Jia C, Feng H and Huang L. 2020. Adaptive regulation of virulence genes by microRNA-like RNAs in Valsa mali. The New phytologist 227(3): 899-913. https://doi.org/10.1111/nph.16561 [ Links ]
Xu M, Li G, Guo Y, Gao Y, Zhu L, Liu Z, Tian R, Ga, C, Han P, Wang N, Guo F, Bao J, Jia C, Feng H and Huang L. 2022. A fungal microRNA-like RNA subverts host immunity and facilitates pathogen infection by silencing two host receptor-like kinase genes. The New phytologist 233(6): 2503-2519. https://doi.org/10.1111/nph.17945 [ Links ]
Zabala-Pardo D, Gaines T, Lamego FP and Avila LA. 2022. RNAi as a tool for weed management: challenges and opportunities. Advance in Weed Science 40(spe1): https://doi.org/10.51694/AdvWeedSci/2022;40:seventy-five006 [ Links ]
Zanini S, Šečić E, Busche T, Galli M, Zheng Y, Kalinowski J and Kogel KH. 2021. Comparative analysis of transcriptome and sRNAs expression patterns in the Brachypodium distachyon- Magnaporthe oryzae pathosystems. International journal of molecular sciences 22(2): 650. https://doi.org/10.3390/ijms22020650 [ Links ]
Zeng J, Gupta VK, Jiang Y, Yang B, Gong L and Zhu H. 2019. Cross-kingdom small rnas among animals, plants and microbes. Cells 8(4): 371. https://doi.org/10.3390/cells8040371 [ Links ]
Zheng Y, Hu Y, Yan S, Zhou H, Song D, Yin M and Shen J. 2019. A polymer/detergent formulation improves dsRNA penetration through the body wall and RNAi-induced mortality in the soybean aphid Aphis glycines. Pest Management Science 75(7): 1993-1999. https://doi.org/10.1002/ps.5313 [ Links ]
Zhou Z, Li X, Liu J, Dongxia H, Chen Q, Liu J, Kong H, Zhang Q, Qianyi Z, Hou D, Zhang L, Zhang G, Liu Y, Zhang Y, Li J, Wang J, Chen X, Wang H, Zhang J, Chen H and Zhang CY. 2015. Honeysuckle-encoded atypical microRNA2911 directly targets influenza A viruses. Cell Research 25: 39-49. https://doi.org/10.1038/cr.2014.130 [ Links ]
Zhou LK, Zhou Z, Jiang XM, Zheng Y, Chen X, Fu Z, Xiao G, Zhang CY, Zhang LK and Yi Y. 2020. Absorbed plant MIR2911 in honeysuckle decoction inhibits SARS-CoV-2 replication and accelerates the negative conversion of infected patients. Cell discovery 6(1): 54. https://doi.org/10.1038/s41421-020-00197-3 [ Links ]
Zhu L, Zhu J, Liu Z, Wang Z, Zhou C and Wang H. 2017. Host-induced gene silencing of rice blast fungus Magnaporthe oryzae pathogenicity genes mediated by the brome mosaic virus. Genes 8(10): 241. https://doi.org/10.3390/genes8100241 [ Links ]
Zotti M, Avila dos Santos E, Cagliari D, Christiaens O, Tizi Taning CN and Smagghe G. 2018. RNA interference technology in crop protection against arthropod pests, pathogens and nematodes. Pest Management Science 74: 1239-1250. https://doi.org/10.1002/ps.4813 [ Links ]
Zotti ., Dos Santos EA, Cagliari D, Christiaens O, Taning CNT and Smagghe G. 2018. RNA interference technology in crop protection against arthropod pests, pathogens and nematodes. Pest Management Science 74: 1239-1250. https://doi.org/10.1002/ps.4813 [ Links ]
Recibido: 30 de Octubre de 2022; Aprobado: 01 de Junio de 2023











 texto en
texto en 


