Introducción
México cuenta con 20 especies del género Persea Mill., de las 85 reportadas en América (Barrientos-Priego et al., 2015). En el sur del Estado de México se distribuyen tres taxones de dos subgéneros: 1) subg. Persea: Persea americana Mill. var. drymifolia (Schltdl. & Cham.) S.F. Blakey, y 2) subg. Eriodaphne (Nees) Meisn.: Persea cinerascens S.F. Blake y Persea hintonii C.K. Allen, conocidos como aguacates y aguacatillos, respectivamente (Kopp, 1966). Durante su evolución, y mediante procesos de hibridación, el aguacate ha generado híbridos naturales que constituyen reservorio de genes hortícolas útiles (Chanderbali et al., 2008). El aguacate tiene su origen en Norteamérica (Gama-Campillo y Gómez-Pompa, 1992; Galindo-Tovar et al., 2007), pero fue domesticado por culturas prehispánicas en Mesoamérica, cuyos nuevos hábitats favorecieron la especiación (Scora et al., 2002), por lo que se han generado nuevos tipos de manera natural, desconocidos, pero con potencial de utilización agronómica.
El presente estudio describe un nuevo tipo de aguacate, localizado en ambiente cálido del Estado de México, México, y semejante, en pubescencia de hoja y tallo joven y grosor de exocarpio, a Persea floccosa Mez (Schroeder, 1951) y a Persea tolimanensis Zentmyer & Schieb. (Schieber y Zentmyer, 1972; 1981). Comparte hábitat con P. americana var. drymifolia, pero difiere de este por presentar hoja pubescente y de mayor tamaño y sin olor a anís, así como fruto de cáscara muy gruesa con lenticelas corchosas (Jaime-Dávila, 2020).
Como hipótesis, la caracterización tanto morfológica como molecular podría apoyar en la identificación del nuevo genotipo y su relación con otras especies, asumiendo que la descripción conjunta podría resultar más eficiente (Rentaría-Alcántara, 2007). Molecularmente se han distinguido híbridos y especies de Persea (Davis et al., 1998; Clegg et al., 1999; Ashworth y Clegg, 2003), y se reconocen RAPD e ISSR como dos marcadores moleculares tradicionales y útiles en la descripción de germoplasma (Fiedler et al., 1998; Reyes-Alemán et al., 2016; Arteaga-Rios et al., 2020). Por lo tanto, el objetivo de este estudio consistió en describir morfológica y molecularmente dos poblaciones del nuevo tipo de aguacate y compararlo con P. floccosa, P. tolimanensis y P. americana var. drymifolia.
Materiales y Métodos
Colecta de material vegetal
Se colectaron 19 muestras de material vegetal: las correspondientes a Persea sp. se colectaron en Zumpahuacán (6) e Ixtapan del Oro (7) y las de P. americana var. drymifolia en Tenancingo (Centro Universitario Tenancingo (CUT) de la Universidad Autónoma del Estado de México (UAEM)) (6). Tres muestras de las especies de referencia P. tolimanensis (2) y P. floccosa (1) fueron tomadas del banco de germoplasma de la Fundación Salvador Sánchez Colín-CICTAMEX, S.C. ubicado en Coatepec Harinas, México (Fig. 1, Cuadro 1).
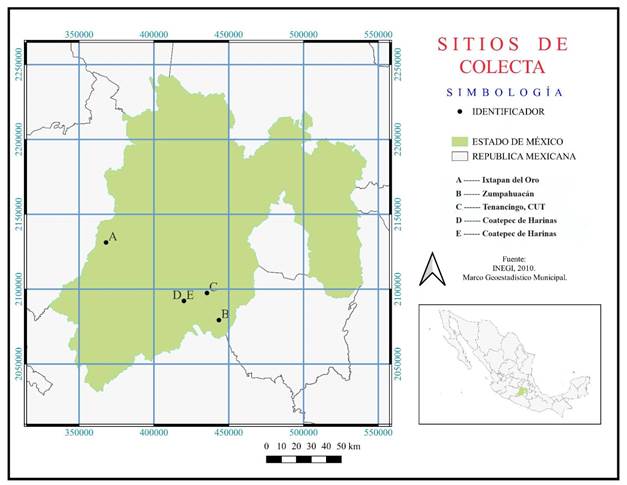
Figura 1: Distribución de colectas, de A. Persea sp. Ixtapan del Oro; B. Persea sp. Zumpahuacán; C. Persea americana var. drymifolia (Schltdl. & Cham.) S.F. Blakey; D. Persea floccosa Mez; E. Persea tolimanensis Zentmyer & Schieb. Mapa de INEGI (2010) modificado en el programa QGIS Development Team v. 3.22 (QGIS Development Team, 2023), por Kenya Ibeth Carreño González y María Eugenia Valdez Pérez.
Cuadro 1: Material vegetal colectado en el Estado de México, México. Colectas empleadas en el estudio morfológico (†), colectas empleadas en el estudio molecular (¶). Los ejemplares están depositados en el herbario CODAGEM (Acrónimo según Thiers, 2024).
| Clave de colecta | Sitio de colecta | Coordenadas altitud | Voucher | Atributos principales | Taxon/raza |
| CUT-M 1†,¶ | Tenancingo, Méx. | 18°58'6.43''N 99°36'46.92''O 2067 m | F. Flores-Ayala y J.C. Reyes Alemán U1 | Fruto pequeño, cáscara delgada | Persea americana Mill. var. drymifolia (Schltdl. & Cham.) S.F. Blakey |
| CUT-M 2†,¶ | Tenancingo, Méx. | 18°58'5.89''N 99°36'45.12''O 2067 m | F. Flores-Ayala y J.C. Reyes Alemán U2 | Semilla grande, escasa inflorescencia | Persea americana Mill. var. drymifolia (Schltdl. & Cham.) S.F. Blakey |
| CUT-M 3†,¶ | Tenancingo, Méx. | 18°58'6.27''N 99°36'46.52''O 2066 m | F. Flores-Ayala y J.C. Reyes Alemán U3 | Fruto mediano, semilla ovada | Persea americana Mill. var. drymifolia (Schltdl. & Cham.) S.F. Blakey |
| CUT-M 5†,¶ | Tenancingo, Méx. | 18°58'6.44''N 99°36'47.02''O 2066 m | F. Flores-Ayala y J.C. Reyes Alemán U5 | Fruto grande, pulpa cremosa | Persea americana Mill. var. drymifolia (Schltdl. & Cham.) S.F. Blakey |
| CUT-M 7†,¶ | Tenancingo, Méx. | 18°58'6.44''N 99°36'46.45''O 2066 m | F. Flores-Ayala y J.C. Reyes Alemán U7 | Fruto pequeño brillante | Persea americana Mill. var. drymifolia (Schltdl. & Cham.) S.F. Blakey |
| CUT-M 10† | Tenancingo, Méx. | 18°58'6.06''N 99°36'45.31''O 2066 m | F. Flores-Ayala y J.C. Reyes Alemán U10 | Brote juvenil muy pubescente | Persea americana Mill. var. drymifolia (Schltdl. & Cham.) S.F. Blakey |
| PERLA-1†,¶ | Zumpahuacán, Méx. | 18°48'25.66''N 99°33'14.05''O 1849 m | L.M. Vázquez-García et al. Z1 | Hoja pubescencia intermedia | Persea sp. Híbrido nuevo |
| PERLA-2†,¶ | Zumpahuacán, Méx. | 18°48'21.80''N 99°33'15.21''O 1833 m | L.M. Vázquez-García et al. Z2 | Fruto pequeño, hoja pubescente | Persea sp. Híbrido nuevo |
| SANPEGU†,¶ | Zumpahuacán, Méx. | 18°49'16.36''N 99°32'19.01''O 1790 m | L.M. Vázquez-García et al. Z3 | Hoja grande ligero olor a anís | Persea sp. Híbrido nuevo |
| SANPEGU-2†,¶ | Zumpahuacán, Méx. | 18°49'16.16''N 99°32'18.52''O 1789 m | L.M. Vázquez-García et al. Z4 | Hoja grande, ligero olor a anís | Persea sp. Híbrido nuevo |
| SANDESP†,¶ | Zumpahuacán, Méx. | 18°48'18.71''N 99°32'12.17''O 1836 m | L.M. Vázquez-García et al. Z5 | Fruto cáscara gruesa, hojas grandes | Persea sp. Híbrido nuevo |
| ZUMP-IGLESIA† | Zumpahuacán, Méx. | 18°49'24.05''N 99°32'14.39''O 1745 m | L.M. Vázquez-García et al. Z6 | Hoja muy oscura, ligero olor a anís | Persea sp. Híbrido nuevo |
| BARR†,¶ | Ixtapan del Oro, Méx. | 19°16'12.35''N 100°15'21.47''O 1698 m | J.C. Jaime-Dávila y J.C. Reyes Alemán I1 | Fruto cáscara gruesa, hojas grandes pubescentes | Persea sp. Híbrido nuevo |
| BARR-2†,¶ | Ixtapan del Oro, Méx. | 19°16'3.11''N 100°15'17.68''O 1725 m | J.C. Jaime-Dávila y J.C. Reyes Alemán I2 | Hojas muy grandes pubescentes | Persea sp. Híbrido nuevo |
| BARR-3† | Ixtapan del Oro, Méx. | 19°16'1.44''N 100°15'18.85''O 1722 m | J.C. Jaime-Dávila y J.C. Reyes Alemán I3 | Hojas muy grandes, ligero olor a anís | Persea sp. Híbrido nuevo |
| SANMAN†,¶ | Ixtapan del Oro, Méx. | 19°16'23.23''N 100°15'34.90''O 1759 m | J.C. Jaime-Dávila y J.C. Reyes Alemán I4 | Fruto mediano de cáscara ligeramente delgada | Persea sp. Híbrido nuevo |
| CASV†,¶ | Ixtapan del Oro, Méx. | 19°16'20.10''N 100°15'21.10''O 1702 m | J.C. Jaime-Dávila y J.C. Reyes Alemán I5 | Hojas grandes pubescentes | Persea sp. Híbrido nuevo |
| CASTI-1†,¶ | Ixtapan del Oro, Méx. | 19°16'25.53''N 100°15'57.12''O 1810 m | J.C. Jaime-Dávila y J.C. Reyes Alemán I6 | Hojas grandes ligeramente torcidas | Persea sp. Híbrido nuevo |
| CASTI-2† | Ixtapan del Oro, Méx. | 19°16'25.86''N 100°15'56.15''O 1815 m | J.C. Jaime-Dávila y J.C. Reyes Alemán I7 | Hojas intermedias, ligero olor a anís | Persea sp. Híbrido nuevo |
| Persea floccosa †,¶ | Coatepec Harinas, Méx. | 18°55'12.23''N 99°45'33.45''O 2214 m | J.C. Jaime-Dávila y J.C. Reyes Alemán C1 | Fruto mediano, cáscara delgada, hoja pequeña muy pubescente | Persea floccosa Mez |
| Persea tolimanensis-1†,¶ | Coatepec Harinas, Méx. | 18°55'14.00''N 99°45'31.91''O 2206 m | J.C. Jaime-Dávila y J.C. Reyes Alemán C2 | Fruto redondo, cáscara muy gruesa, sabor semi amargo | Persea tolimanensis Zentmyer & Schieb. |
| Persea tolimanensis-2†,¶ | Coatepec Harinas, Méx. | 18°55'14.03''N 99°45'32.04''O 2206 m | J.C. Jaime-Dávila y J.C. Reyes Alemán C3 | Fruto redondo, cáscara muy gruesa, sabor semi amargo | Persea tolimanensis Zentmyer & Schieb. |
El material vegetal se colectó en noviembre de 2018 y de enero a marzo de 2019, consistiendo en tallos jóvenes, hojas e inflorescencias de árboles in situ (Fig. 2). Cada colecta consistió en 10 ramificaciones vegetativas de 30 cm de longitud, 10 panículas florales y 10 frutos maduros por orientación cardinal del árbol. Las muestras se colocaron en bolsas plásticas y se trasladaron en una hielera de 54 qt capacidad (Coleman®, modelo 5295A780G, Wichita, EUA) al laboratorio del Centro Universitario Tenancingo (UAEM), se refrigeraron para su conservación y enseguida se realizaron las mediciones descritas. Los ejemplares fueron depositados en el herbario CODAGEM (Acrónimo según Thiers, 2024) de la Facultad de Ciencias Agrícolas de la Universidad Autónoma del Estado de México.
Caracterización morfológica
Las mediciones morfológicas se hicieron con vernier digital Stainless Steel 2020® (Sunrom, Gujarat, India), regla de 30 cm y balanza granataria OHAUS 1650-W0® (Global Test Supply, North Carolina, EUA), de acuerdo con los siguientes descriptores: International Plant Genetic Resources Institute (IPGRI, 1995), International Union for the Protection of New Varieties of Plants (UPOV, 2006) y Barrientos-Priego et al. (2010). Estadísticamente la unidad experimental consistió en un árbol como ejemplar único por accesión (Cuadro 1). Se generaron 10 repeticiones por característica: de hoja, flor y fruto por lado cardinal del árbol. Sesenta y cuatro características morfológicas fueron consideradas: 19 cuantitativas y 45 cualitativas (Cuadro 2). Se integraron tres matrices básicas de datos: hoja, flor y fruto (Material suplementario 1, 2, 3).
Cuadro 2: Características morfológicas utilizadas y su unidad de medición. †=con base en descriptores para aguacate (Persea spp.) (IPGRI, 1995). ¶=con apoyo en el manual gráfico para la descripción varietal de aguacate (Barrientos-Priego et al., 2010). §=con apoyo en “Guidelines for the conduct of test distinctness, uniformity and stability” (UPOV, 2006).
| Características | Hoja | Flor | Fruto |
| Cuantitativas | 1 Longitud de hoja (cm)† | 1 Longitud de eje floral (cm)† | 1 Longitud de fruto (cm)† |
| 2 Ancho de hoja (cm)¶,§ | 2 Número de ramificaciones por inflorescencia† | 2 Diámetro de fruto (cm)† | |
| 3 Relación longitud × ancho de hoja (cm)¶,§ | 3 Número de flores por inflorescencia† | 3 Relación longitud/diámetro de fruto¶,§ | |
| 4 Longitud de peciolo (cm)¶ | 4 Longitud de pedicelo (cm)† | 4 Diámetro de inserción de pedicelo (cm)¶,§ | |
| 5 Número de venas secundarias¶,§ | 5 Longitud de pétalos (cm)† | 5 Longitud de pedicelo (cm)† | |
| 6 Ancho de pétalos (cm) | 6 Grosor de la cáscara (cm)† | ||
| 7 Longitud de sépalos (cm)† | |||
| 8 Ancho de sépalos (cm) | |||
| Cualitativas | 6 Forma de la hoja† 1=ovado, 2=estrechamente obovado, 3=obovado, 4=oval, 5=casi redondo, 6=cordiforme, 7=lanceolado, 8=oblongo, 9=oblongo-lanceolado, 10=otro | 9 Color de lenticelas¶,§ 1=verde, 2=rojo | 7 Forma del pedicelo† 1=cilíndrico, 2=cónico, 3=redondeado, 4=otro |
| 7 Forma del ápice†: 1=muy agudo, 3=agudo, 5=intermedio, 7=obtuso, 9=muy obtuso | 10 Tipo de floración† 1=tipo A, 2=tipo B, 3=desconocido | 8 Presencia de cuello¶,§,Þ 0=ausente, 1=presente | |
| 8 Torcedura de la hoja¶,§,Þ 0=ausente, 1=presente | 11 Tipo de nectario¶,§ 1=sésil, 2=pedunculado | 9 Depresión en el extremo peduncular¶,§,Þ 0=ausente, 1=presente | |
| 9 Torcedura del ápice¶,§,Þ 0=ausente, 1=presente | 12 Presencia de polen†,Þ 0=ausente, 1=presente | 10 Posición de pedicelo en fruto† 1=central, 2=asimétrico, 3=muy asimétrico, 4=extremadamente asimétrico | |
| 10 Margen de la hoja (ondulación)†,Þ 1=entera, 2=ondulada | 13 Posición Estilo de flor† 1=derecho, 2=torcido, 3=otro | 11 Forma del ápice del fruto† 1=profundamente deprimido, 2=ligeramente deprimido, 3=aplanado, 4=redondeado, 5=puntiagudo | |
| 11 Relieve de las venas superiores† 3=hundido, 5=intermedio, 7=elevada | 14 Pubescencia del sépalo interno¶,§,Þ 0=ausente, 1=presente | 12 Conspicuidad de lenticelas¶,§ 1=inconspicuo o débil, 2=medio, 3=fuerte | |
| 12 Pubescencia en el envés† 34 | 15 Densidad de pubescencia del sépalo†,¶,Þ | 13 Tamaño de lenticelas† 3=pequeño, 5=intermedio, 7=grande | |
| 13 Olor a anís† 3=débil, 5=intermedio, 7=fuerte | 16 Abundancia de la floración | 14 Color de lenticelas¶,§,Þ 1=crema, 2=amarillo, 3=verde claro, 4=marrón | |
| 17 Posición de la inflorescencia† 1=terminal, 2=subterminal, 3=axilar, 4=otra | 15 Brillantes de fruto¶,§,Þ 1=ausente o débil, 2=medio, 3=fuerte | ||
| 18 Pubescencia del pétalo 0=ausente, 1=presente | 16 Superficie de fruto†3=suave, 5=intermedio, 7=áspero | ||
| Cualitativas | 19 Densidad de pubescencia del pétalo† 3=escasa, 5=intermedia, 7=densa | 17 Persistencia de perianto† 0=ausente, 1=presente | |
| 18 Grosor del pedicelo en comparación con el pedúnculo¶,§,Þ 1=igual, 2=mayor | |||
| 19 Forma de pedicelo† 1=cilíndrico, 2=cónico, 3=redondeado, 4=otro | |||
| 20 Color de pedicelo† 1=amarillo, 2=verde, 3=naranja, 4=rojo, 5=café, 6=otro | |||
| 21 Superficie de pedicelo¶,§,Þ 1=lisa, 2=rugosa | |||
| 22 Color de fruto maduro† 1=ligeramente verde, 2=verde, 3=verde obscuro, 4=amarillo, 5=rojo, 6=morado, 7=negro, 8=moteado, 9=otro | |||
| 23 Consistencia de la cáscara¶,§ 1=membranosa, 2=coriácea, 3=corchosa | |||
| 24 Adherencia de la cáscara a la pulpa† 3=leve, 5=intermedia, 7=fuerte | |||
| 25 Color principal de la pulpa¶,§,Þ 1=blanquecino, 2=crema, 3=amarillo, 4=verde claro | |||
| 26 Color junto a la cáscara¶,§,Þ 1=verde claro, 2=verde medio, 3=verde amarillo. | |||
| 27 Ancho de la capa junto a la cáscara¶,Þ 3=estrecho, 5=medio, 7=ancho | |||
| 28 Fibra en la pulpa† 3=bajo, 5=intermedio, 7=alto | |||
| 29 Textura de la pulpa† 1=acuoso, 2=mantecoso, 3=pastosa, 4=granular, 5=otro | |||
| 30 Aroma a anís de la pulpa¶,§,Þ 0=ausente, 1=presente | |||
| 31 Relación longitud de fruto/ longitud de semilla¶,§,Þ 1=muy pequeña, 3=pequeña, 5=media, 7=grande, 9=muy grande | |||
| 32 Pedicelo con cabeza de clavo† 0=ausente, 1=presente |
Análisis estadísticos de datos morfológicos
Para diferenciar morfológicamente a los genotipos, se utilizaron la técnica de conglomerados, el método jerárquico, el criterio de clasificación Average Linkage, la distancia euclídea, estandarización de datos, y los caracteres morfológicos asociados a hoja, flor y fruto de los genotipos colectados (Cuadros 1 y 2). Los caracteres fueron el criterio de número mínimo y con la distancia media, se seleccionaron los conglomerados. El análisis de discriminantes lineal se usó para clasificar individuos en colectas e identificar variables asociadas a la clasificación (Johnson, 2000; Balzarini et al., 2008). Ambos procedimientos se realizaron con las rutinas de Info Stat v. 2017 (Di Rienzo et al., 2017).
Caracterización molecular
El ADN genómico se extrajo de 100 mg de tejido fresco de hojas tiernas de tallos jóvenes de 5 a 10 cm colectadas por genotipo, distintas a las utilizadas para la descripción morfológica. Se colocaron en hielera y transportaron en cadena fría al laboratorio ya mencionado, para su proceso. Se utilizó el kit FAST ID Genomic DNA Extraction®. La concentración de ADN en las muestras se midió mediante espectrofotómetro ND-1000 Nano Drop (Thermo Fisher Scientific®, Waltham, EUA). El cociente de medición fotométrica, DO260/280, del ADN varió entre 1.9 y 6.2, que indica calidad aceptable de ADN genómico (Riepl et al., 2011). El rendimiento de ADN en 100 mg de tejido de hoja joven fue de 73.2 ng µl-1, en promedio.
La mezcla de PCR para los iniciadores RAPD contenía 2 µl de ADN (20 ng µl-1), 10 µl de MyTaq Mix, 2 µl de iniciador y 4 µl de H2O destilada-desionizada libre de nucleasas (SIGMA™), para un volumen final de 28 µl. Los mismos componentes se usaron para ISSR. La PCR se llevó a cabo en un termociclador GENE Amp PCR System 2700® (Applied Biosystems®, Foster City, EUA) con el siguiente programa: para RAPD: 1 min a 94 °C, seguido de 35 ciclos a 94 °C × 30 s, 40 °C × 30 s y 72 °C × 1.5 min, con una extensión final de 72°C × 2.5 min. Para ISSR: 1 min a 94 °C, seguido de 38 ciclos a 94 °C × 30 s, 48 °C × 30 s y 72 °C × 2 min con una extensión final de 72 °C × 2.5 min.
Los iniciadores utilizados y los índices de eficiencia obtenidos se presentan en el Cuadro 3. Los productos de PCR se corrieron en electroforesis en una Celda MS CREEN Cleaver Scientific® (Warwick, UK) con geles de agarosa entre 0.9 y 1.2%, con una corriente de 90 a 120 V × 2 a 3 horas. La selección de oligonucleótidos informativos se basó en estudios previos con especies del género Persea (Reyes-Alemán et al., 2013; 2016).
Cuadro 3: Tipo de marcador molecular utilizado, secuencia del iniciador, bandas de ADN × locus, número de amplicones, porcentaje de contribución y bandas polimórficas por iniciador, contenido de información polimórfica (PIC), poder de resolución (RP) e índice del marcador (IM) obtenidos. 1Compañia Sigma-Aldrich, *B=G, T, C; D=G, A, T; Y=C, T; R=A, G, iniciadores anclados, **iniciador sin ancla.
| Tipo de marcador | Iniciador | Secuencias (5’-3’) | Locus × bandas DNA | Amplicones/ iniciador | % Contribución | Bandas polimórficas presentes/ esperadas | PIC | RP | IM |
| RAPD1 | A 15 | TTC CGA ACC C | 17 | 83 | 5.4 | 83/83 | 0.36 | 1.33 | 29.9 |
| C 08 | TGG ACC GGT G | 13 | 244 | 15.8 | 208/244 | 0.43 | 14.08 | 89.4 | |
| C 10 | TGT CTG GGT G | 7 | 157 | 10.17 | 139/157 | 0.44 | 4.08 | 61.1 | |
| C 13 | AAG CCT CGT C | 10 | 189 | 12.25 | 189/189 | 0.50 | 8.33 | 94.5 | |
| E 7 | AGA TGC AGC C | 10 | 207 | 13.4 | 189/207 | 0.48 | 8.33 | 90.7 | |
| E 14 | TGC GGC TGA G | 5 | 190 | 12.3 | 100/190 | 0.47 | 2.08 | 47 | |
| E 16 | GGT GAC TGT G | 14 | 268 | 17.4 | 268/268 | 0.49 | 16.33 | 131.3 | |
| E 18 | GGA CTG CAG A | 11 | 205 | 13.3 | 205/205 | 0.50 | 10.08 | 102.5 | |
| Total | 87 | 1543 | 100 | 1381/1583 | 3.67 | 64.64 | - | ||
| Promedio | 10.87 | 192.9 | 12.5 | 172.6/192.9 | 0.45 | 8.08 | 80.8 | ||
| ISSR* | AC(GACA)₄ | ACGACAGACAGACAGACA | 7 | 255 | 21.14 | 129/255 | 0.49 | 4.08 | 63.21 |
| DBDA (CA)₇ | DBDACACACACACACACA | 6 | 115 | 9.54 | 115/115 | 0.50 | 3 | 57.5 | |
| (AC)₈YG | ACACACACACACACACYG | 8 | 214 | 17.74 | 160/214 | 0.50 | 5.33 | 80 | |
| (GA)₈YC | GAGAGAGAGAGAGAGAYC | 6 | 141 | 11.7 | 123/141 | 0.50 | 3 | 61.5 | |
| (TCC)₅RY | TCCTCCTCCTCCTCCRY | 13 | 289 | 23.96 | 253/289 | 0.46 | 14.08 | 116.4 | |
| (GACA)₄** | GACAGACAGACAGACA | 8 | 192 | 15.92 | 156/192 | 0.49 | 5.33 | 76.44 | |
| Total | 48 | 1206 | 100 | 936/1206 | 2.93 | 34.83 | - | ||
| Promedio | 8 | 201 | 16.7 | 156/201 | 0.49 | 5.81 | 75.8 |
Análisis estadístico de datos moleculares
Con los productos amplificados se integró una matriz conjunta de caracteres binarios en donde las bandas polimórficas se codificaron con 1 y su ausencia con 0. La información se procesó mediante el software Free Tree v. 0.91.5 (PavlíČek et al., 1999) y se visualizó mediante el programa TreeView v. 1.6.6. (Page, 2001). Para RAPD se utilizó el criterio de distancia de similitud de Jaccard (Jaccard, 1908) y el método de construcción del árbol Neighbor-Joining (Saitou y Nei, 1987). Para ISSR se utilizó el mismo criterio de distancia de similitud anterior, pero se empleó el método de agrupamiento UPGMA por resultar más informativo (Sneath y Sokal, 1973). Se realizó un bootstrap de 1000 repeticiones en cada caso.
La eficiencia de los iniciadores se basó en el contenido de información polimórfica (PIC), poder de resolución (RP) e índice del marcador (IM) (Cuadro 3). El PIC se determinó mediante la ecuación (1) descrita por Roldán-Ruiz et al. (2000):
Donde PICi, es el contenido de información polimórfica, fi es la frecuencia de las bandas presentes y 1-fi, la frecuencia de las bandas ausentes.
La habilidad de los primers más informativos se basó en su poder de resolución (RP). Fue calculado mediante la fórmula (2) descrita por Prevost y Wilkinson (1999), que se refiere a la correlación entre oligonucleótidos con habilidad para distinguir entre especies:
Donde lb, representa la información de la banda calculado mediante la fórmula (3):
Donde p, corresponde a la proporción de todos los genotipos que contienen a la banda l.
El índice del marcador (MI) determina la utilidad total de un marcador molecular, mide el equilibrio entre el polimorfismo detectado y la medida de polimorfismos múltiples en un ensayo (Varshney et al., 2007; Arteaga Rios, 2017).
Donde PIC, es el contenido de información polimórfica, y el número de bandas polimórficas, las contenidas en el sistema RAPD o ISSR respectivamente.
Resultados
Descripción morfológica del nuevo genotipo
TIPO: CASV: MÉXICO. Estado de México, Valle de Bravo, municipio Ixtapan del Oro, San Martín Ocoxochitepec, 1702 m, 19°16'20.10"N, 100°15'21.10"O, selva baja caducifolia barranca, 20.I.2019, J. C. Reyes Alemán 5 (holotipo: CODAGEM!, isotipo: por distribuirse).
Árbol de crecimiento ascendente, 12-20 m de altura, alternancia productiva, longevo, 80 años o más, brotes nuevos de color verde claro; hojas maduras forma oval u obovoide, densamente pubescentes, 16-20 cm de largo, 10-14 cm de ancho, sin olor a anís al estrujarse; hábito floral tipo A, raquis floral joven con lenticelas verdes, pubescencia interna de tépalos moderada a abundante, estilo corto pubescente, nectario sésil; fruto con exocarpio grueso, coriácea, corchosa, 1.2-2 mm de grosor, forma aperada o piriforme, cuello 11-16 cm de largo × 7 cm de diámetro, color de maduración púrpura a negro de la parte estilar al ápice, pulpa amarillo verdoso, consistencia acuosa o aceitosa, fibras conspicuas; semilla con débil adherencia de pulpa a la semilla, ovada, grande con relación al fruto (Fig. 2A,B).
Fenología: florece en febrero y fructifica de marzo a abril, logra 11 a 12 meses de flor a fruto.
Distribución: se encuentra en zona de transición bosque mesófilo, bosque de Pinus-Quercus, selva baja caducifolia y pastizal, clima templado-semicálido, subhúmedo; 1700-2000 m.s.n.m., con topografía accidentada (Caballero-Martínez et al., 2009; Emeterio-Lara et al., 2016).
Vegetativamente las características discriminantes para la hoja del nuevo genotipo fueron mayor longitud, forma ovada, borde ondulado, densidad de pubescencia en envés, semejante con P. floccosa y P. tolimanensis. Una mayor densidad de pubescencia distingue a P. floccosa, ondulación y relieve elevada de venas a P. tolimanensis. Por el contrario, P. americana var. drymifolia se distingue por su hoja angosta, lanceolada, glabra, con doblamiento del ápice y olor a anís (Fig. 3). El nuevo genotipo se distinguió por presentar el mayor tamaño de hoja, de 16 a 37.5 cm de longitud, con respecto a P. americana var. drymifolia (raza mexicana). También por presentar una densidad de tricomas (pubescencia) de media a densa y presencia de ondulación en el borde de la hoja, de débil a media, semejante a P. tolimanensis y P. floccosa (Fig. 3).
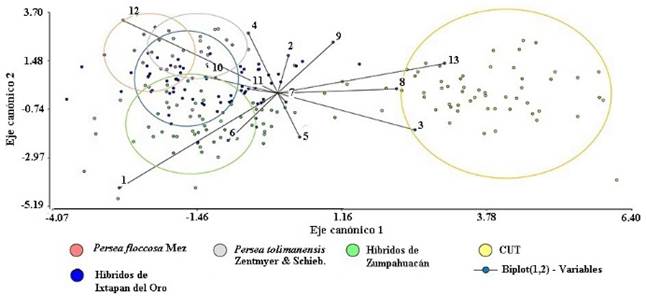
Figura 3: Análisis discriminante de hoja de 22 genotipos con 13 variables y cinco agrupamientos, de acuerdo con las características previamente descritas en el Cuadro 2. CUT=Centro Universitario Tenancingo.
La variabilidad encontrada en la inflorescencia del nuevo genotipo destacó la presencia de pubescencia muy densa al interior del sépalo, pedicelo más largo, pétalo más ancho y mayor número de ramificaciones por inflorescencia (Fig. 4A,B). Por el contrario, en P. americana var. drymifolia predominó un menor número de flores por inflorescencia, pubescencia más escasa en pétalo y sépalo más angosto. Así mismo, la abundante floración del nuevo genotipo difiere de P. floccosa por presentar una mayor ramificación y número de flores y de P. tolimanensis por presentar un nectario más visible y mayor cantidad de polen.
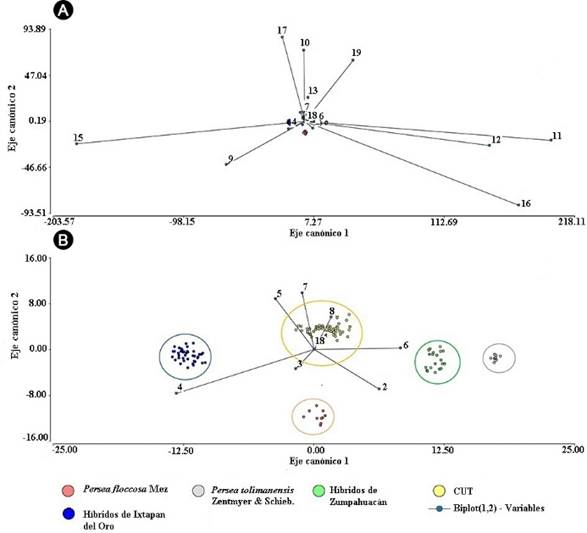
Figura 4: Análisis discriminante de inflorescencias de 14 genotipos con 19 variables y cinco agrupamientos de acuerdo con las características previamente descritas en el Cuadro 2. CUT=Centro Universitario Tenancingo.
En la localidad de Ixtapan del Oro la floración del nuevo genotipo presentó menos densidad de pubescencia de pétalo y una longitud de pedicelo más corta. En el caso de Zumpahuacán, el nuevo genotipo se caracterizó por mayor número de ramificaciones, pétalo más ancho y un nectario pedunculado. En todos los casos P. americana var. drymifolia se distinguió por tener menor número de flores por inflorescencia, sépalo más angosto y pubescencia del pétalo ausente (Fig. 4A). El nuevo genotipo de Zumpahuacán fue semejante con P. tolimanensis, en un estilo de flor más erecto, presencia de nectario, mucho más pubescencia y anchura de pétalo.
La distinción más importante del nuevo genotipo es su exocarpio de fruto grueso, de forma redonda a aperada, diámetro pronunciado, grandes lenticelas corchosas, pedicelo cabeza de clavo, pulpa pastosa adherida a la semilla y semilla grande (Fig. 5). Persea tolimanensis con la cual es semejante, se distingue por su fruto más redondo, achatado, exocarpio grueso, forma irregular, áspero, verde obscuro, de sabor amargo, exocarpio duro cuando madura, y su ramificación comienza en lo alto del tronco (Scora et al., 2002). En el caso del fruto de Persea floccosa, este es menos largo que ancho, lenticelas corchosas menos notorias, sus tallos tiernos y hojas son muy pubescentes y flocosas (Scora et al., 2002).
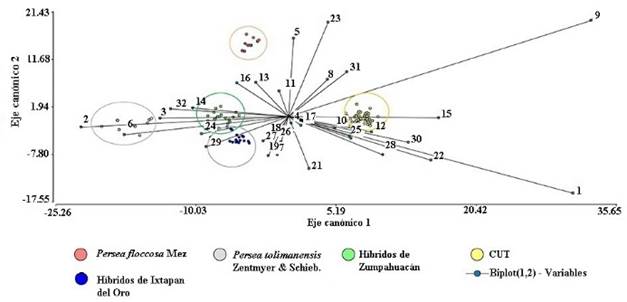
Figura 5: Análisis discriminante de fruto de 12 colectas con 32 variables y cinco agrupamientos de acuerdo con las características previamente descritas en el Cuadro 2. CUT=Centro Universitario Tenancingo.
Los frutos del nuevo genotipo de Zumpahuacán fueron semejantes con P. tolimanensis en el grosor del exocarpio (0.9-2.2 mm) y diámetro del fruto (5.73 a 7.90 cm). Los de Ixtapan del Oro fueron distintos por la consistencia seca de su pulpa. En comparación, en P. americana var. drymifolia (CUT), en el que sobresale su longitud, posición central del pedicelo, depresión en su extremo peduncular, conspicuidad de lenticelas y fibras, brillantez, color de exocarpio negro a rojizo, delgado y pulpa con aroma a anís (Fig. 5).
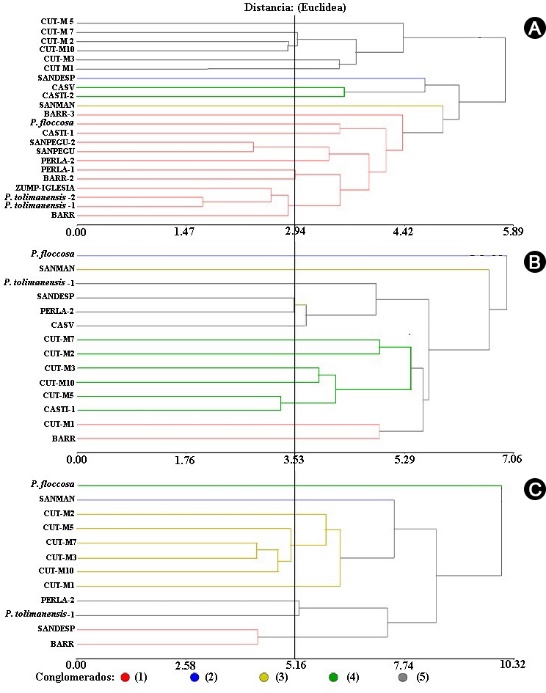
Con una distancia Euclídea de 2.92, 3.53 y 5.16 en hoja, flor y fruto, respectivamente (Fig. 6A), se distinguió que los genotipos de Zumpahuacán e Ixtapan del Oro tienen una hoja más grande, redonda y pubescente, excepto el genotipo SANMAN, en comparación con P. americana var. drymifolia. Este último se caracteriza por su típica hoja aguda, lanceolada y olor a anís. En floración, sobresale la mayor longitud de sépalos y pétalos en P. tolimanensis y P. floccosa que la raza mexicana. El genotipo de raza mexicana CUT-M1 sobresale en pubescencia de tépalos, al igual que BARR, SANMAN y P. floccosa. (Fig. 6B). En fruto, el genotipo SANMAN fue semejante, en forma aperada, lenticelas corchosas y cáscara verde en comparación con P. floccosa, siendo estas características típicas de este último. Los genotipos PERLA-2, SANDESP y BARR de Zumpahuacán son semejantes en grosor de exocarpio, redondez, lenticelas corchosas y consistencia de pulpa a P. tolimanensis (Fig. 6C).
Caracterización molecular
El poder de resolución en RAPD fue de 64.64 y en ISSR de 34.83. Para el primero, cinco de ocho oligonucleótidos fueron los más informativos con 11.43 promedio individual, los tres menos informativos de 7.49 en conjunto, y la media colectiva fue de 8.08. En ISSR, tres de seis iniciadores sumaron 24.74 de RP, los menos informativos 10.08, y una media colectiva de 5.81. Solo la mitad fue contundentemente informativa. Lo anterior expresa un diagnóstico completo del perfil genético acorde con Gilbert et al. (1999).
El ADN utilizado tuvo un cociente fotométrico OD260/280, de 1.96 a 6.26 unidades, que es considerado de calidad de acuerdo con Riepl et al. (2011). Sus productos de PCR tuvieron un tamaño de 250 a 900 pb, 99% de polimorfismos y 1417 bandas en el sistema RAPD, y de 200 a 1000 pb con 917 bandas en ISSR. Los iniciadores que aportaron mejor Información Polimórfica (PIC) al sistema RAPD para la identificación molecular fueron A15, E18 y C13, con 0.36 a 0.50 de PIC. En tanto, (TCC)₅RY y (AC)₈YG, DBDA(CA)₇ y (GA)₈YC lo hicieron en ISSR con 0.46 y 0.50, respectivamente. Mediante A15 y E16 de RAPD, se logró un poder de resolución (RP) de 64.64 puntos con variaciones de 1.33 a 16.33, respectivamente. En tanto con ISSR fue de 14.08 a 34.83 con (TCC)₅RY, DBDA(CA)₇ y (GA)₈YC, respectivamente.
Relaciones genéticas y similitud entre colectas
Se generó un árbol filogenético mediante Jaccard (1908) y Neighbor-joining (Saitou y Nei, 1987) para RAPD. Se lograron cuatro agrupamientos del nuevo genotipo, distintos a P. tolimanensis y P. floccosa. Solo SANMAN, CASTI-1 y CASV fueron afines. Los grupos se relacionan con P. americana var. drymifolia (Fig. 7). Un segundo árbol mediante Jaccard (1908) y UPGMA (Sneath y Sokal, 1973) para ISSR confirmó separación entre el nuevo genotipo y P. tolimanensis y P. floccosa (Fig. 8).
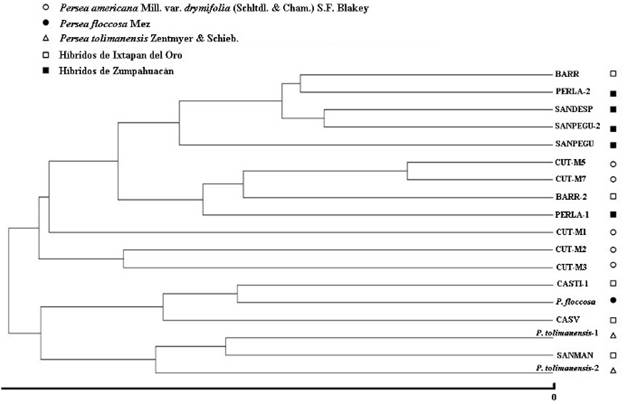
Figura 7: Filograma de 18 colectas de Persea Mill. generado mediante ocho iniciadores RAPD: se utilizaron el índice Jaccard y el método Neighbor-joining con un bootstrap de 1000 repeticiones.

Figura 8: Filograma de 18 colectas de Persea Mill. generado mediante seis iniciadores ISSR: se utilizaron el índice Jaccard y el método UPGMA con un bootstrap de 1000 repeticiones.
Con base en los marcadores utilizados, se sugiere hibridación del nuevo genotipo con P. americana var. drymifolia, y una relación directa con P. tolimanensis y P. floccosa, de seis individuos del nuevo genotipo: SANMAN, CASTI-1, CASV, BARR3, SANDESP y PERLA. RAPD, además de relacionar a P. floccosa y P. tolimanensis con seis individuos del nuevo genotipo, sugiriendo hibridación de este con P. americana var. drymifolia.
ISSR separó a los nuevos genotipos y a tres ejemplares de P. americana var. drymifolia, de P. floccosa y de P. tolimanensis. (Fig. 8). La relación genética de CASTI-1, CASV, SANMAN, BARR3, SANDESP y PERLA con P. floccosa y P. tolimanensis mediante RAPD es coherente con la similitud morfológica.
Discusión
Vegetativamente los individuos del nuevo genotipo difieren de las especies de referencia. Sin embargo, sobresale en los individuos la densidad de pubescencia en envés de la hoja, tamaño mayor de hoja, ausencia de olor a anís con elevado relieve en venas, que son características semejantes a las presentes en P. floccosa y P. tolimanensis. El tipo y densidad del indumento en hoja y estructuras reproductivas en Lauraceae delimita especies, siendo una característica constante, pero variable en Persea. Van der Werff (2002) cita ejemplos de especies variables en pubescencia: Persea bullata L.E. Kopp de Ecuador y Persea boldufolia Mez de Perú, con plantas glabras, moderadamente pubescentes y densamente pubescentes.
En floración los ejemplares del nuevo genotipo son variables, pero destaca su abundante floración, sépalo pubescente, pétalo ancho, mayor ramificación de inflorescencia y flores con pedicelo más largo, en comparación con P. floccosa y P. tolimanensis. Es distintivo que en Persea sus tépalos son iguales o desiguales, deciduos en fruto maduro, anteras cuadriloculares, glándula estipitada (nectario) en la base de los estambres de la tercera hilera y pistilo pubescente (van der Werff, 2002). Sin embargo, su variación constituye criterio insuficiente para separar especies (van der Werff, 2002), por lo que este autor considera a las especies citadas solo formas botánicas de P. americana. El genotipo de estudio difiere de P. americana var. drymifolia tan solo por mayor pubescencia en pétalo, sépalo más ancho y más número de flores, carácter que limita sugerir una nueva especie.
En fruto se encontró variabilidad, pero sobre todo sobresale el exocarpio grueso, forma redonda a aperada, lenticelas grandes, corchosas, semilla grande, pulpa con fibra y poca palatabilidad. Difiere de P. tolimanensis porque en este el fruto es más redondo, de sabor amargo, y de P. floccosa, debido a que en este su fruto es menos largo que ancho, lenticelas menos notorias con hojas más pubescentes (Scora et al., 2002), pero semejantes en grosor de exocarpio. El grosor de exocarpio en Persea apoya la definición entre especies (Scora et al., 2002). Persea floccosa es considerado filogenéticamente afín a P. americana var. drymifolia (Furnier et al., 1990), pero este último tiene exocarpio delgado de fruto, brillante, lenticelas conspicuas y pulpa de color verde con fibra, de acuerdo con el análisis discriminante (Fig. 5).
El nuevo genotipo presenta la siguiente combinación de caracteres: exocarpio grueso, hoja pubescente con olor a anís, algunos con fruto típico de raza mexicana y hoja ancha pubescente. Lo anterior intuye que solo hubo hibridación de P. americana var. drymifolia, como lo sugiere van der Werff (2002), y que sean solo variantes de P. americana.
La variabilidad de P. americana le confiere ser considerada especie polimórfica, afín con Persea schiedeana Nees, de pedicelos más cortos (8 mm de largo), hojas más angostas, brácteas de yemas, brotes y hojas uniformemente más pubescentes. Persea americana var. drymifolia, altamente distribuida en el centro de México (Damián-Nava et al., 2017), ha sido nombrada de distinta manera de acuerdo con van der Werff (2002) en su sinopsis de Persea (Lauraceae) en América central: Persea americana var. angustifolia Miranda, identificada en Puebla, México; Persea drymifolia Schltdl. & Cham. 1, colectada en la cadena montañosa del centro y sur de México; Persea gratissima var. drymifolia (Schltdl. & Cham.) Mez, identificada en Orizaba, Veracruz, México; Persea edulis Raf., relacionado con Persea americana Mill.; Persea floccosa Mez, identificada en México, Puebla; Persea gigantea L.O. Williams, identificada en Honduras; Persea gratissima C.F. Gaertn., relacionado con Persea americana Mill.; Persea gratissima var. oblonga Meisn., identificada en México, en Perú por Ruiz y Pavon, y en Brasil; Persea gratissima var. praecox Nees; Persea gratissima var. macrophylla Meisn.; Persea leiogyne Blake; Persea nubigena L.O. Williams; Persea americana var. nubigena (L.O. Williams) L.E. Kopp; Persea paucitriplinervia Lundell, y Persea steyermarkii C.K. Allen, como evidencia del polimorfismo de P. americana, y descrito a través del tiempo.
Algunos autores indican que Persea tienen poca combinación entre sus especies, no así al interior de cada una, y destaca el carácter monofilético del género como sugiere Rohwer et al. (2009), razón que justifica la hibridación observada dentro de P. americana var. drymifolia en el estudio. Por el contrario, basado en secuenciación de nucleótidos en P. americana var. drymifolia, reveló una baja recombinación genética de esta, pese a alogamia, pero exaltó a la mutación como una ruta para generar su diversidad (Chen et al., 2008). La var. drymifolia mostró desviación en el equilibrio Hardy-Weinberg y desequilibrio de ligamiento (Guzmán et al., 2017); es decir, la hibridación entre razas tiene dificultad, pero no así entre la misma raza, razón por la que la raza mexicana, o var. drymifolia, presenta gran variabilidad y fácilmente genera polimorfismos, como lo observamos en los genotipos de. estudio.
Chen et al. (2008) analizaron genéticamente las variedades drymifolia, guatemalensis (P. americana var. guatemalensis (L.O. Williams) Scora) y americana (P. americana var. americana), conocidas como las razas mexicana, guatemalteca y antillana, respectivamente, y encontraron desequilibrio de ligamiento, contrario al analizarse por separado. Lo anterior apoya que la preservación genética y morfológica que el aguacate de estudio ha tenido, se debe a su confinamiento geográfico y a su poca atracción por parte del humano, lo que lo ha aislado por tiempo considerable y conducido a hibridación natural a partir de P. americana.
Con respecto a su semejanza con ciertas especies de P. americana y acorde con Ashworth y Clegg (2003), la hibridación del nuevo aguacate pudo haber iniciado muy recientemente, ya que tiene pocas diferencias genéticas y morfológicas. Por el contrario, la hibridación entre P. americana var. drymifolia y var. guatemalensis sucedió durante un periodo prolongado, según Williams (1977). Gama-Campillo y Gómez-Pompa (1992) definieron que P. americana aún es una especie semi-domesticada, en recombinación continua por intercambio constante de material genético, lo que sugiere que el nuevo aguacate es un híbrido de Persea americana var. drymifolia con P. tolimanensis o P. floccosa, aún sin evidencia de tratarse de una nueva especie.
El entrecruzamiento es común entre el género Persea, y ha sido fomentado por su hábito de reproducción cruzada conocida como dicogamia protogínica. Esta es responsable de la gran variabilidad y similitud entre especies que ha conducido a la hibridación natural del aguacate, ya que si bien las especies del género Persea poseen dificultades genéticas para cruzarse entre ellas, como se ha sustentado, no ha sido una limitante como se evidencia en este estudio. Las relaciones filogenéticas que guardan las especies del género Persea son evidentes; ya que en algún momento de su evolución se han relacionado, como argumenta van der Werff (2002), compartiendo un origen monofilético (Rohwer et al., 2009), solo se han separado y se han confinado debido a eventos climáticos, geográficos y/o evolutivos.
Un estudio reciente ha revelado que el germoplasma del aguacate colombiano no pertenece a ninguna de las tres razas clásicas de aguacate mesoamericano, si no a un nuevo acervo genético de P. americana exclusivo de la región andina y que no había sido reportado hasta la fecha. Este gran hallazgo fortalece la hipótesis de la hibridación natural. Los autores lo confirman como un nuevo linaje de aguacate que divergió a partir de la raza antillana durante el Pleistoceno, y refieren que está relacionado con dos antiguos grupos genéticos más antiguos que las clásicas razas reconocidas hasta el momento (Berdugo‑Cely et al., 2023).
Boza et al. (2018) asumieron que existe una compleja mezcla interracial en el género Persea, de acuerdo con un estudio reciente de marcadores moleculares SSR realizado mediante 354 colectas de P. americana, P. nubigena, P. krugii Mez y P. schiedeana. Así mismo, el estudio reciente del genoma del aguacate mexicano (Persea americana var. drymifolia) ha revelado un ejemplo de profundas relaciones filogenéticas entre las angiospermas, y ha mostrado que existe una hibridación constante, así como la habilidad de adaptación del aguacate ante patógenos (Rendón-Anaya et al., 2019). Molecularmente se robusteció la descripción en Persea, como ha sido documentado por Rodríguez-Medina et al. (2009); Rohwer et al. (2009). Los marcadores RAPD e ISSR revelaron que existe gran variabilidad entre genotipos. Acorde con Gilbert et al. (1999), se infirieron relaciones genéticas importantes entre los genotipos de estudio, de acuerdo con los filogramas obtenidos (Figs. 7, 8). Aunque también se sugiere que ha existido erosión genética entre los genotipos al haber generado pocos polimorfismos.
La caracterización molecular de los recursos genéticos del aguacate es tan importante, debido a que optimiza su utilización al identificar similitudes y variaciones desconocidas entre las colectas, que permite establecer colecciones de referencia y conduce a fomentar las tareas del mejoramiento genético del cultivo, como sugieren Berdugo‑Cely et al. (2023). Si la tarea del mejoramiento pudiera ser llevada a cabo tomando como base genética a los genotipos nativos nuevos del centro de México, como los del presente estudio, eso sería el aporte de la presente investigación.
Los iniciadores utilizados reportaron arriba de 50% de polimorfismos obtenidos (Cuadro 3). En RAPD, E16 tuvo la contribución más alta al sistema (17.4%), y A15 la más baja (5.4%), siendo congruentes con el número de amplicones y bandas polimórficas obtenidas. El índice descriptivo PIC fue mayor de 0.25 en los ocho iniciadores. Esto indica que fueron altamente informativos, validando dicho sistema, donde C13, E18 y E16 tuvieron el contenido de información polimórfica más alto, aun cuando su porcentaje de contribución no fue el mayor. Por no detectar mayor número de bandas polimórficas, su valor discriminante fue alto y semejante a lo reportado por Arteaga-Rios et al. (2020).
En ISSR, la contribución mayor al sistema fue de (TCC)₅RY (23.96%), y la más baja DBDA (CA)₇ (9.54%), siendo congruentes con su número de amplicones y polimorfismos reportados. El porcentaje de información polimórfica (PIC) en los seis iniciadores, al ser mayores de 0.25 y cercanos a 0.5, indican que fueron altamente informativos aun con un porcentaje de contribución menor, lo cual muestra que poseen un valor discriminante alto.
El poder de resolución (RP) en RAPD evidencia que los iniciadores E16, E18 y C08 lograron el valor más alto: 16.33, 10.08 y 14.08, respectivamente, congruente con el índice del marcador (IM) reportado: 131.3, 102.5 y 89.4 respectivamente. Sin embargo, debido a que RP se basa en el índice de información que ofrece cada una de las bandas (Ib) bajo una condición óptima hipotética (50% de los genotipos conteniendo una banda), e IM se basa en PIC, obtenido este de los valores reales, en ambos parámetros, tanto en el poder de resolución (RP) como en el índice del marcador (IM), influye de forma directa el número de bandas polimórficas obtenidas. Por lo tanto, aunque el iniciador C13 obtuvo un PIC elevado, su RP e IM no fueron tan altos, comparado con E16 y E18. En ISSR se observó un comportamiento semejante, habiendo sido (TCC)₅RY el iniciador que presentó el mayor número de polimorfismos obtenidos con un RP de 14.08, y un IM de 116.4. Los resultados fueron semejantes con lo reportado por Arteaga-Rios et al. (2020).
RAPD e ISSR separaron en dos grupos (Zumpahuacán e Ixtapan del Oro) a los ejemplares del nuevo tipo de aguacate; probablemente estos fueron desplazados durante el pasado, en el supuesto de la existencia de grupos geográficos, intactos, por altitud y latitud (Chen et al., 2009; Guzmán et al., 2017), o por afinidad filogenética con la especie que los originó, en este caso con P. floccosa, P. tolimanensis y P. americana. Se destaca la convivencia con el subgénero Eriodaphne acorde con Rohwer et al. (2009), puesto que convive con P. cinerascens y P. hintonii Allen. Se sugiere una composición genética afín entre colectas (Figs. 7, 8). En el supuesto de que una baja cantidad de individuos brinda un menor despliegue de bandas, hay más representatividad de los individuos contrastados, se representan todas las bandas posibles y se encuentran alelos poco frecuentes más fácilmente (Gilbert et al., 1999). Los ejemplares se separaron de las especies de referencia de forma congruente, por localidad (Figs. 7, 8).
Chanderbali et al. (2013) mencionaron que P. americana, distribuida en Mesoamérica, es una especie polimórfica con controversias taxonómicas, desde la clasificación de Kopp (1966). Popenoe (1935) indicó que todos los aguacates cultivados son formas hortícolas de P. americana y sus prototipos silvestres, formas geográficas, relacionados genéticamente (Fiedler et al., 1998). Mediante RFLP, Furnier et al. (1990) separaron a P. nubigena, P. steyermarkii y P. floccosa de P. americana, y concluyeron que son tan solo variantes. Ashworth y Clegg (2003) consideraron que las razas de P. americana se domesticaron por separado. Gama-Campillo y Gómez-Pompa (1992) mostraron evidencia de la introgresión racial desde culturas prehispánicas. El nuevo aguacate podría ser una forma silvestre con introgresión reciente de P americana.
El aguacate fue domesticado por dos causas principales: la intervención humana y la hibridación natural (Gama-Campillo y Gómez-Pompa, 1992; Galindo-Tovar et al., 2007). En el primer caso se genera un cuello de botella donde solo las características de interés se seleccionan: tamaño de fruto, tiempo de maduración, sabor, etc. (Chen et al., 2008), excluyendo los caracteres poco atractivos. En el segundo caso se da paso al proceso de hibridación natural cuando el aguacate, como el de este estudio, es segregado al no ser atractivo.
Conclusiones
El nuevo tipo de aguacate comparte hábitat con Persea americana var. drymifolia de quien procede, siendo un ejemplo de hibridación natural constante. Es relacionado morfológica y molecularmente con P. tolimanensis y P. floccosa, y podría considerarse variedad botánica nueva de P. americana, confirmando su carácter polimórfico.











 nueva página del texto (beta)
nueva página del texto (beta)