Introducción
El bocachico Prochilodus magdalenae (Steindachner, 1879) constituye uno de los principales recursos pesqueros del país, representando el 30% de la captura total en la cuenca del río Magdalena en el año 2010. Sin embargo, la abundancia del recurso ha disminuido a causa de los numerosos impactos antropogénicos, dentro de los que se destacan: la contaminación, la sedimentación de los ríos, la deforestación de las zonas ribereñas, la construcción de hidroeléctricas (Atencio, 2000; Monteiro et al., 2006; Marrugo-Negrete et al., 2008) y la sobrepesca (Valderrama et al., 2002; Agostinho et al., 2005; Hatanaka et al., 2006), causando en las poblaciones naturales fragmentación, reducción de su hábitat, interrupción del proceso de migración y disminución de la tasa de renovación poblacional (Hatanaka et al., 2006).
Dentro de las medidas pesqueras utilizadas para la mitigación del impacto generado sobre la población de bocachico están las tradicionales como son: la implementación de tallas mínimas de captura (25 cm), la prohibición de algunas artes de pesca y el repoblamiento (Agostinho et al., 2005). Siendo evidente la degradación de los ambientes acuáticos, las medidas preventivas para la conservación del bocachico deben ser tomadas en cuanto al banco genético in situ de la población (Campos, 2009). En este sentido, el repoblamiento constituye la estrategia más utilizada para la recuperación pesquera de P. magdalenae. Sin embargo, su ineficiente utilización puede acarrear la pérdida de la diversidad genética, convirtiéndose en una amenaza para las poblaciones naturales (Povh, 2007). Las grandes e irreversibles alteraciones ambientales en la cuenca del río Magdalena, guían a una perspectiva de manejo en cautividad de las especies en peligro o que presentan algún grado de vulnerabilidad como el bocachico (Mojica et al., 2012). Para lo cual es necesario establecer grupos de reproductores mantenidos en estaciones piscícolas, a través de una selección de individuos que representen la diversidad genética de las poblaciones naturales (Ryder, 1986; Seal, 1988; Wasko et al., 2004) y que al momento de la liberación de las progenies, estas contribuyan al mantenimiento y/o aumento de la variabilidad genética de la población natural. Por eso la correcta selección del grupo de reproductores que sean usados para este propósito, es de suma importancia (Wasko et al., 2004).
Actualmente, los programas de repoblamiento que se llevan a cabo en Colombia no se basan en criterios técnicos y científicos, ya que las granjas piscícolas no implementan criterios genéticos en sus programas de cruza, favoreciendo la mezcla de reproductores emparentados, que en consecuencia reducen la variabilidad genética (Moreira et al., 2003; Povh et al., 2006). Además, promueven en la población mayor sensibilidad a las variaciones ambientales, afectando el crecimiento y la reproducción (Moreira et al., 2001; Povh et al., 2006), y en ocasiones provocan la extinción de una especie (Lopera-Barrero et al., 2006).
Identificar los reproductores con las mejores condiciones genéticas para producir semilla con fines de repoblamiento y analizar la calidad genética de las larvas producidas, es el procedimiento pertinente para corroborar que se está proporcionando la información adecuada para la recuperación genética de una población en el medio natural (Machado-Schiaffino, 2007). En este sentido, el objetivo de la presente investigación fue evaluar el efecto de la variabilidad genética individual de los reproductores en la producción de larvas de bocachico (Prochilodus magdalenae) con fines de repoblamiento.
Materiales y métodos
El estudio fue realizado en el Centro Acuícola y Agroindustrial de Gaira (74° 07´ W y 11° 11´ N), el cual se encuentra bajo la dirección del SENA Regional Magdalena (Colombia), localizado a 7 Km de la ciudad de Santa Marta, con un área de 5 ha y una altitud promedio de 4 m.s.n.m. Este centro posee 25 estanques, donde son mantenidos los reproductores de bocachico obtenidos del medio natural, los cuales fueron marcados con microchips (transponders) y caracterizados genéticamente por Muñoz (2013). La caracterización de estos reproductores se realizó a través de la genotificación de siete loci microsatélites propuestos por Rueda et al. (2011) y cuyo objetivo final consistió en establecer un sistema de reproductores con criterio genético para obtener progenies de P. magdalenae con fines de repoblamiento (Muñoz, 2013).
La parte experimental se llevó a cabo entre los meses de septiembre y octubre de 2012, coincidiendo el segundo periodo de maduración de los reproductores de bocachico en la cuenca del Magdalena (Jiménez-Segura et al., 2010). Los animales seleccionados para los diferentes ensayos estuvieron entre los 12 a 16 meses de edad y no habían sido empleados para eventos reproductivos (Muñoz, 2013).
Del grupo de reproductores caracterizados previamente por Muñoz (2013) , fueron seleccionadas nueve hembras y 18 machos con las siguientes características: tres hembras y seis machos con heterocigosidad individual menor o igual a 0.3, tres hembras y seis machos con heterocigosidad individual entre 0.3 y 0.6, y por último fueron seleccionadas tres hembras y seis machos con heterocigosidad individual mayor a 0.6. La heterocigosidad individual fue calculada con la información obtenida de Muñoz (2013), por medio de la relación entre el número de loci para los cuales los organismos manifestaron ser heterocigotos y el número total de loci analizados. De igual manera, se tuvo en cuenta el estado de maduración de los animales seleccionados por medio del análisis presuntivo de las características externas. En hembras su estado de maduración se verificó a través del abdomen abultado y la papila genital dilatada, mientras que los machos maduros fueron reconocidos por espermiación constante. Para los intervalos de heterocigosidad individual, cada hembra fue colocada en una pileta de reproducción en proporción 2:1, en la cual se realizó la inducción hormonal con Extracto Pituitario de Carpa (EPC) a razón de 6.6 mg/Kg. En hembras se aplicaron dos dosis en intervalos de 12 horas, entre cada aplicación. La primera dosis equivalente al 10%, y la segunda al 90% (Atencio, 2001). En machos se aplicó una sola dosis correspondiente al 10% aplicada con la segunda dosis de las hembras. El desove ocurrió entre seis y siete horas después de la inducción hormonal y se llevó a cabo de manera semi-natural, mediante la expulsión espontánea de los gametos en las piletas de reproducción y la fertilización fue natural (Atencio, 2001).
Se midieron las variables de fecundidad, tasa de fertilización, tasa de eclosión y supervivencia. La fecundidad (número de huevos desovados por hembra) se calculó por volumetría después del desove. El promedio de la tasa de fertilización se calculó cinco horas post-fertilización mediante muestreos al azar de un número superior a 100 huevos. Fueron considerados como huevos viables aquellos que presentaban un aspecto transparente. El valor promedio obtenido en el análisis de los 100 huevos fue extrapolado, al número total de huevos de la muestra. La tasa de eclosión se midió 12 horas post-fertilización, aplicando el mismo método muestreal. La supervivencia de las larvas fue medida por volumetría, 12 horas post-eclosión (Atencio, 2000). Durante la realización de los experimentos de inducción e incubación, se midieron diferentes parámetros fisicoquímicos del agua como la temperatura, el oxígeno disuelto, el pH con un equipo multiparámetros WTW 340i. Adicionalmente se monitoreó la dureza del agua y el amonio con un kit hach.
Para la toma de muestras para realizar las pruebas genéticas, se fijaron 30 larvas en etanol al 96% por cada experimento realizado, 12 horas post-eclosión. Estas larvas fueron almacenadas en tubos Eppendorf de 1.5 mL a una temperatura de 15 °C para su uso posterior.
La extracción de ADN genómico de las larvas de P. magdalenae, se realizó utilizando el kit para extracción de ADN MasterPure(tm) DNA Purification (Epicentre Biotechnologies(r)) conforme a las instrucciones del fabricante. Se verificó la obtención y calidad del ADN empleando electroforesis horizontal, corrida a 80 voltios durante 40 minutos, en geles de agarosa al 0.8% y tinción con bromuro de etidio. La visualización del gel se realizó en un fotodocumentador Biodoc UVP LLC con transiluminador de luz ultravioleta. La cantidad de ADN extraído fue cuantificada a través de espectrofotometría de luz UV, la cual osciló entre 50 y 200 ng/µl. El ADN fue conservado a -20 °C.
La variabilidad genética fue analizada usando siete loci microsatélites específicos de Prochilodus lineatus (Valenciennes, 1837), cuya amplificación cruzada fue probada exitosamente en P. magdalenae (Rueda et al., 2011). Las amplificaciones fueron realizadas en un termociclador ESCO-SWIF MaxPro empleando un volumen de reacción final de 10 µl conteniendo 200 µM de dNTPs, 1X PCR buffer, 0.2 µM de cada primer (Forward y Reverse), 2 mM de MgCl2 y 0.25 U de Taq polimerasa. Las condiciones de amplificación fueron: desnaturalización inicial a 94 °C por 5 min, seguido de 30 ciclos a 94 °C (30s), temperatura de alineamiento de cada primer (50-62 °C) durante (30 s), 72 °C (30 s), y una extensión final a 72 °C durante 10 min. Los productos obtenidos de las amplificaciones, fueron analizados por medio de un equipo de electroforesis capilar de alta resolución QIAxcel Advance System (QIAGEN), utilizando un kit de alta resolución (QIAxcel DNA High Resolution Kit) y un marcador de peso de concentraciones conocidas (DNA Size Marker 50-800 bp v2.0 QIAGEN), el cual permitió cuantificar el tamaño o peso en pares de bases (pb) de cada banda y así distinguir los individuos homocigotos de los heterocigotos.
Para evaluar el efecto de la variabilidad genética individual de los reproductores sobre peso, fecundidad, tasa de fertilización, tasa de eclosión y supervivencia de larvas de P. magdalenae, se realizó un análisis de varianza a una vía (ANOVA) comparando cada una de estas variables de manera individual entre los diferentes intervalos de heterocigosidad seleccionados para el estudio. Estas comparaciones fueron realizadas usando el programa estadístico IBM SPSS Statictics v.20 (IBM Corp. Released, 2011). Previo a estas comparaciones fueron comprobados los supuestos de normalidad y homogeneidad de varianzas de cada una de las variables usando este mismo programa. Se realizó la prueba de Tukey cuando fueron encontradas diferencias significativas entre los niveles de heterocigosidad individual de los reproductores para cada variable seleccionada.
El efecto de la variabilidad genética individual de los reproductores, sobre la calidad genética de las progenies de P. magdalenae, fue evaluada calculando la heterocigosidad observada (Ho), heterocigosidad esperada (He) y el índice de endogamia (Fis) de las larvas, por medio del paquete computacional GENETIX v.4.05 (Belkhir, 2003). Con los valores de los parámetros de Ho, He y Fis obtenidos para los reproductores (Muñoz, 2013) y para las progenies producidas, se realizaron comparaciones de estos parámetros entre cada intervalo de heterocigosidad individual (0-0.3; 0.3-0.6; y mayor a 0.6), a través de un análisis de varianza (ANOVA), con la previa comprobación de los supuestos de normalidad y homogeneidad de varianzas, usando el programa estadístico IBM SPSS Statictics v.20 (IBM Corp. Released, 2011). Se realizó la prueba de Tukey para observar grupos homogéneos dentro de los datos obtenidos.
Resultados
Efecto de la variabilidad genética individual de los reproductores sobre la producción de progenies de bocachico con fines de repoblamiento. Los valores promedio de peso de los reproductores, fecundidad, tasa de fertilización, tasa de eclosión y supervivencia, se muestran en la Tabla 1. Para las variables peso, tasa de fertilización y supervivencia, se encontraron diferencias significativas (p < 0.05) entre el primer intervalo de heterocigosidad individual más bajo (0-0.3) y los intervalos de heterocigosidad individual comprendidos entre 0.3-0.6 y 0.6-1. Mientras que, el número de huevos por hembra (tasa de fecundidad) y la tasa de eclosión no presentaron diferencias significativas (p > 0.05) para los tres niveles de heterocigosidad individual analizados.
Tabla 1: Valores de los parámetros productivos de peso de los reproductores, fecundidad, tasa de fertilización, tasa de eclosión y supervivencia de larvas de bocachico (Prochilodus magdalenae) obtenidas por desove semi-natural (valor promedio ± d.e).

Los parámetros fisicoquímicos para este experimento no presentaron variaciones significativas durante la fase de inducción al desove y la fase de incubación de los huevos, manteniéndose constantes a lo largo del experimento, garantizando así las condiciones óptimas para la fertilización y eclosión de los huevos en los diferentes tratamientos (Tabla 2).
Tabla 2: Valores de los parámetros fisicoquímicos (temperatura, pH, dureza y amonio) medidos durante la inducción de los reproductores y la incubación de los huevos de bocachico (Prochilodus magdalenae; valor promedio ± d.e).

Efecto de la variabilidad genética individual de los reproductores sobre la calidad genética de progenies producidas de bocachico con fines de repoblamiento. Los valores de heterocigosidad observada (Ho), heterocigosidad esperada (He) y el índice de endogamia (Fis) de los reproductores utilizados para la obtención de las progenies, presentaron diferencias significativas (p < 0.05) entre los tres intervalos de heterocigosidad individual (Tabla 3). Mientras que, en las progenies obtenidas sólo se presentaron diferencias significativas (p < 0.05) en los parámetros Ho y Fis entre los dos primeros intervalos de heterocigosidad individual (0-0.3 y 0.3-0.6) con respecto al último (0.6-1). Para el caso de la He, no se presentaron diferencias significativas entre los tres intervalos de heterocigosidad individual (Tabla 4).
Tabla 3: Valores de heterocigosidad observada (Ho), heterocigosidad esperada (He) e índice de endogamia (Fis) de los reproductores de bocachico (Prochilodus magdalenae) utilizados para la obtención de las progenies analizadas (valor promedio ± d.e, n=3).
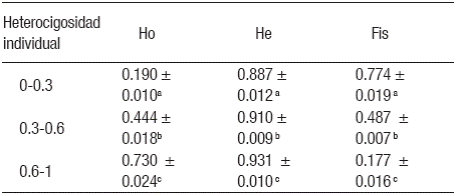
Letras diferentes dentro de una misma columna indica diferencias significativas (p < 0.05).
Tabla 4: Valores de heterocigosidad observada (Ho), heterocigosidad esperada (He) e índice de endogamia (Fis) de las progenies de bocachico (Prochilodus magdalenae) obtenidas para los tres intervalos de heterocigosidad individual (valor promedio ± d.e, n=3).
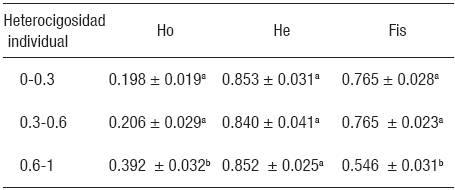
Letras diferentes dentro de una misma columna indica diferencias significativas (p < 0.05).
Por su parte, los valores de heterocigosidad observada (Ho), heterocigosidad esperada (He) y el índice de endogamia (Fis), disminuyeron de reproductores a progenies para los tres intervalos de heterocigosidad individual (Tablas 3-4).
Discusión
El mantenimiento de altos niveles de variabilidad genética y una baja endogamia, es el principal objetivo en los programas de conservación, en los cuales la diversidad alélica puede ser usada como un indicador para este propósito (Koljonen, 1989). Sin embargo, a pesar de la importancia que reviste el mantenimiento de la variabilidad genética en una población, es común encontrar información donde se documenta que los lotes de reproductores mantenidos en cautiverio en las estaciones piscícolas, han sufrido una pérdida significativa de la variabilidad genética (Machado-Schiaffino et al., 2007; Muñoz, 2013). Esto es debido al manejo reproductivo llevado a cabo, ya que dado que se desconocen las relaciones genéticas de los reproductores usados, individuos estrechamente relacionados pueden ser cruzados para la obtención de nuevas progenies (Kang et al., 2006). De acuerdo con lo anterior, varios autores proponen la evaluación de la variabilidad genética de los reproductores y las progenies mantenidas en cautiverio, como requisito fundamental en los programas de repoblamiento (Moreira, 2001; Porta et al., 2006; Povh et al., 2008). En este estudio se comprueba que la variabilidad genética individual de los reproductores de Prochilodus magdalenae utilizados en programas de repoblamiento, es una condición que afecta los parámetros de producción de los reproductores y la calidad genética de sus progenies. En este sentido, se pudo observar que la tasa de fertilización y la supervivencia, son parámetros reproductivos condicionados por el nivel de heterocigosidad individual de los reproductores. Es probable que la dominancia reproductiva o hipótesis de competencia espermática (Sivinski, 1984) también afecte a dichos parámetros de producción cuando se usan sistemas reproductivos semi-naturales, ya que la dominancia de algunos machos (supuestamente los más fuertes y con mejores características reproductivas) en la fertilización de los óvulos, puede estar influenciada por la variabilidad genética individual del reproductor. Sin embargo, debido a la falta de información no es posible hacer conjeturas ni sacar conclusiones al respecto de esta hipótesis.
Para la calidad genética de las progenies obtenidas a través de los diferentes niveles de heterocigosidad individual, el nivel más alto (> 0.6) les proporcionó a las progenies una mayor heterocigosidad observada (Ho) y una menor endogamia (Fis), evidenciando que la condición genética de las larvas es dependiente de la condición genética de los reproductores usados. A pesar de los bajos valores de variabilidad genética encontrados para las progenies, en el presente estudio se demuestra como con el conocimiento de las características genéticas y definiendo cruces específicos entre los reproductores, es posible mantener un manejo eficiente de los mismos, incrementando poco a poco la variabilidad genética de las progenies que serán usadas en los programas de repoblamiento (Lopera-Barrero et al., 2008). Esto contradice a lo propuesto por algunos autores como Wasko et al. (2004) y Sekino et al. (2004), donde se asume que la pérdida de variabilidad genética en estaciones piscícolas es normalmente irreversible. Sin embargo, en poblaciones naturales la pérdida de variabilidad genética, es recuperada lentamente por mutación o migración (Avise et al., 1987), mientras que en poblaciones cautivas se podría realizar con una renovación del plantel de reproductores o el establecimiento de un sistema de reproductores con criterio genético. Por lo tanto, un manejo genético adecuado de los peces mantenidos en cautiverio, es un paso importante para aumentar el éxito de los programas de repoblamiento realizados en el hábitat acuático.
Los bajos valores de variabilidad genética obtenidos en los cruces de individuos con baja variabilidad individual (0-0.3 nivel de heterocigosidad), pueden ser producto del cruzamiento entre reproductores genéticamente emparentados, lo que puede aumentar la homocigosis, reducir la variabilidad genética de las progenies y tener efectos significativos de depresión endogámica (Lopera-Barrero et al., 2007). Sumado a lo anterior, la disminución encontrada en la variable de supervivencia pudo también influir en los bajos valores de heterocigosidad observada en algunos intervalos de variabilidad genética (Machado-Schiaffino et al., 2007). Sin embargo, el parámetro Ho fue mayor para los reproductores contrastados con los encontrados en las progenies. Rodríguez et al. (2010), registraron para Brycon orbignyanus (Valenciennes, 1850) una tendencia a la disminución de la variabilidad genética durante las fases de crecimiento, ya que la heterocigosidad observada fue disminuyendo desde el reproductor, siguiendo con las larvas (0.864) hasta alevinos de 30 días (0.635), por la desaparición de alelos específicos en la población y posiblemente un factor que afectó de manera significativa fue la mortalidad en esta etapa. En el presente estudio se observó una disminución de la variabilidad genética de reproductores a larvas, evidenciándose la necesidad de ampliar el estudio hasta los 30 días de crecimiento de las progenies.
Por otra parte, en el estudio realizado por Wasko et al. (2004), en Brycon cephalus (Günther, 1869), encontraron que el cruce de individuos emparentados provocó una disminución de la variabilidad genética de las progenies a lo largo del análisis de tres generaciones. En consecuencia, la pérdida de heterocigosidad tiene un fuerte efecto en la depresión endogámica, principalmente en la adaptación, capacidad reproductiva, eficiencia fisiológica (Povh et al., 2008; Lopera-Barrero et al., 2010) y en la expresión de alelos deletéreos, lo que conduce a una importante disminución poblacional que puede tener consecuencias como el colapso de la población y posteriormente la extinción (Leuzzi et al., 2004). Por lo tanto, la variabilidad genética, es importante para la persistencia evolutiva de las especies. Los intentos para la rehabilitación de poblaciones en peligro, depende de la manutención de la variabilidad genética de las poblaciones silvestres en sistemas artificiales a través de la implementación de criaderos y lotes de reproductores para tal fin (Allendorf et al., 1986). Es de vital importancia la manera cómo se conforma un lote de reproductores utilizados para la producción de larvas usadas en los programas de conservación, ya que la equivocada selección de éstos, incrementa el riesgo de aparición de cuellos de botella en la población (Machado-Schiaffino et al., 2007) además de un aumento de la endogamia de las progenies (Rodríguez et al., 2010). Los resultados obtenidos en el presente estudio, son una herramienta importante para el manejo reproductivo y genético de la población de Prochilodus magdalenae usada en los programas de repoblamiento. En este sentido, es importante que durante la realización de los cruzamientos destinados para la producción de larvas, sean usados individuos con alta variabilidad genética individual, debido a que estos garantizarán el incremento en la heterocigosidad observada de las progenies y así preservar la variabilidad genética de la población. De igual forma, es recomendable que se realice un seguimiento de dicha variabilidad a través del tiempo, el cual va a ofrecer un amplio conocimiento de la información gentética de la nueva generación. De acuerdo a lo anterior, dicho monitoreo debe realizarse entre los 60 y 90 días, para determinar objetivamente la variabilidad genética resultante de los cruces (variabilidad genética de los individuos a ser liberados) y así determinar la verdadera viabilidad del repoblamiento (Rodríguez et al., 2010).











 nueva página del texto (beta)
nueva página del texto (beta)


